胰腺癌
胰腺癌是美国第四大癌症死因。它是一种严重的疾病,恶性细胞在胰腺组织中形成。胰腺是一个位于腹部深处的器官,横跨在胃的下部和脊柱前方(图1)。它主要的功能是产生消化液和激素,用于消化和吸收食物以及吸收营养物质。
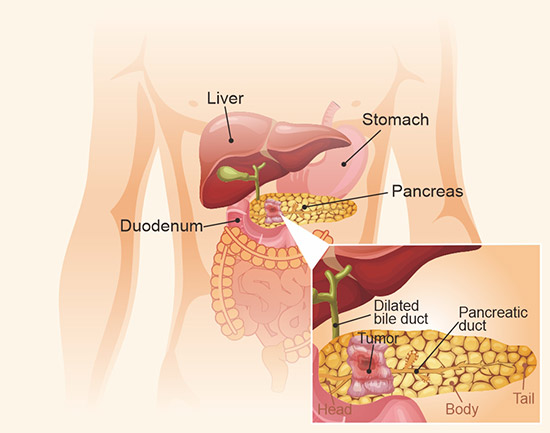
图1. 胰腺结构示意图
*此图表来自JAMA [1] 上发表的文章
胰腺癌是指胰腺细胞在胰腺内发生异常的DNA突变,导致细胞分裂和不受控制的生长,形成肿瘤。胰腺可以发生两种类型的生长,包括恶性和非恶性肿瘤。胰腺中最常见的癌症类型始于胰腺内携带消化酶的管道的细胞(胰导管腺癌)。由于胰腺位于腹腔深处,胰腺癌很难检测和诊断。肿瘤可能引起黄疸、疼痛、食欲减退、胰腺炎或意外的体重减轻等症状。
胰腺癌的分子病理学主要由KRAS激活突变主导,这种突变在超过90%的肿瘤中存在。TP53、CDKN2A和SMAD4的失活突变在50-80%的胰腺癌中出现,而其他基因,包括ARID1A和TGFBR2,在约10%的肿瘤中突变 [2-5]。本文根据NCG提供的信息,列出了与胰腺癌相关的部分靶点。NCG是一个用于分析癌症基因的重复性、同源性和网络特性的网络资源。
在这里,我们展示了胰腺癌分子病理学中涉及的几个关键靶点,包括:
● CDKN2A(cyclin-dependent kinase inhibitor 2A),也称为p16-INK4a、MTS-1或CDK4I,是一个肿瘤抑制基因,通过抑制负责启动G1/S期转变的cyclinD-CDK4和cyclinD-CDK6复合物来调节细胞周期进程。它是癌症中最常见的改变基因之一,在散发性胰腺癌中突变的发生率令人印象深刻,约有98%的病例发生失活 [6] [7]。
● SMAD4(SMAD家族成员4)是一种肿瘤抑制蛋白,在TGFβ家族受体激活后,以异三聚体形式转位到细胞核,激活基因表达并引起生长抑制 [8]。在胰腺导管腺癌中,KRAS、TP53、SMAD4和CDKN2A等四个驱动基因被报道为代表性的与癌症相关基因 [9]。此外,SMAD4的突变在约50%的胰腺癌中发生,大多数导致蛋白质失活。
● ARID1A(AT-rich interactive domain containing protein 1A)是SWI/SNF复合物的一部分,是一个多亚基复合物,通过依赖ATP的方式转移、插入或驱逐核小体来重塑染色质。ARID1A在胰腺导管腺癌中常见突变,并且新兴研究表明,ARID1A以一种区段特异的方式抑制胰腺肿瘤发生。此外,在导管细胞中,这个过程似乎与MYC促进的蛋白质合成有关 [10]。
● KMT2C(Lysine N-methyltransferase 2C),也称为髓系/淋巴系或混合谱系白血病蛋白3,是KMT2家族组蛋白赖氨酸甲基转移酶的成员,包括KMT2A、KMT2B、KMT2C、KMT2D、KMT2E、KMT2F、KMT2G和KMT2H [11]。其中,KMT2C和KMT2D是COMPASS复合物中的单甲基转移酶,两者都以H3K4为靶标进行表观遗传调控 [12]。几项研究观察到在胰腺癌患者中存在KMT2C(错义)突变和KMT2D(错义)突变。此外,根据Sausen等人的报道,KMT2C/D的无义/错义突变与胰腺癌患者的较好预后相关 [13]。
● RNF43(ring finger 43)作为一种跨膜E3泛素连接酶,通过泛素化Wnt受体frizzled来下调Wnt/β-catenin通路 [14]。RNF43的突变已在多种肿瘤中被发现,包括囊性胰腺肿瘤。
参考文献:
[1] Alexandra Moore and Timothy Donahue. Pancreatic Cancer [J]. JAMA. 2019, 322 (14): 1426.
[2] Wang, L. et al. Whole-exome sequencing of human pancreatic cancers and characterization of genomic instability caused by MLH1 haploinsufficiency and complete deficiency [J]. Genome Res. 2012, 22, 208–219.
[3] Waddell, N. et al. Whole genomes redefine the mutational landscape of pancreatic cancer [J]. Nature. 2015, 518, 495–501.
[4] Bailey, P. et al. Genomic analyses identify molecular subtypes of pancreatic cancer [J]. Nature. 2016, 531, 47–52.
[5] Jorg Kleeff, Murray Korc, Minoti Apte et al. Pancreatic cancer [J]. Nat Rev Dis Primers. 2016, 21 (2):16022.
[6] Schutte, M.; Hruban, R.H.; Geradts, J. et al. Abrogation of the RB/p16 tumor-suppressive pathway in virtually all pancreatic carcinomas [J]. Cancer Res. 1997, 57, 3126–3130.
[7] Jonas Cicenas, Kotryna Kvederaviciute, Ingrida Meskinyte et al. KRAS, TP53, CDKN2A, SMAD4, BRCA1, and BRCA2 Mutations in Pancreatic Cancer [J]. Cancers. 2017, 9 (12): 42–.
[8] Cao, D.; Ashfaq, R.; Goggins, M.G. et al. Differential expression of multiple genes in association with MADH4/DPC4/SMAD4 inactivation in pancreatic cancer [J]. Int. J. Clin. Exp. Pathol. 2008, 1, 510–517.
[9] Takahiro Yokose, Minoru Kitago, Sachiko Matsuda et al. Combination of KRAS and SMAD4 mutations in formalin-fixed paraffin-embedded tissues as a biomarker for pancreatic cancer [J]. Cancer Sci. 2020, 111(6):2174-2182.
[10] Sam C Wang, Ibrahim Nassour, Shu Xiao et al. SWI/SNF component ARID1A restrains pancreatic neoplasia formation [J]. Gut. 2019, 68:1259-1270.
[11] Allis CD, Berger SL, Cote J et al. New nomenclature for chromatin-modifying enzymes [J]. Cell. 2007, 131:633–6.
[12] Rokutan, H.; Hosoda, F.; Hama, N. et al. Comprehensive mutation profiling of mucinous gastric carcinoma [J]. J. Pathol. 2016, 240, 137–148.
[13] Sausen, M.; Phallen, J.; Adleff, V. et al. Clinical implications of genomic alterations in the tumour and circulation of pancreatic cancer patients [J]. Nat. Commun. 2015, 6, 7686.
[14] Xiao Yan Chang, Yan Wu, Ying Jiang et al. RNF43 Mutations in IPMN Cases: A Potential Prognostic Factor [J]. Gastroenterology Research and Practice. 2020, 1457452.






