组蛋白磷酸化
在1970年代,研究人员开始在各种生物样本中检测到磷酸化的组蛋白。使用放射性标记技术的实验揭示了组蛋白,尤其是组蛋白H1,可以被磷酸化。在1980年代,研究开始暗示组蛋白磷酸化与染色质结构和功能之间存在联系。在了解组蛋白磷酸化的角色方面,一个重大突破出现在1990年代,当时的研究人员发现了它在细胞周期调控中的作用。到了21世纪,对组蛋白磷酸化的研究显著扩展。
1. 什么是组蛋白磷酸化?
组蛋白磷酸化的概念首次在1967年被提出 [1]。组蛋白磷酸化酶将磷酸基团沉积到组蛋白上的丝氨酸(S)、苏氨酸(T)或酪氨酸(Y)残基上。大多数磷酸化发生在H3S10和H3S28上,以及H3T3、H3T6和H3T11上。组蛋白磷酸化可以根据特定背景和磷酸化位点的位置对基因表达产生不同影响。
2. 参与组蛋白磷酸化的三个基本组成部分
每个四个核心组蛋白都容易被不同的蛋白激酶磷酸化,而磷酸化过程可以被磷酸酶逆转,这个过程被称为去磷酸化。识别和结合磷酸化组蛋白,以及将信号转化为不同生物输出的最终过程是由磷酸化组蛋白读取器完成的。
2.1 蛋白激酶
蛋白激酶,是将磷酸基团添加到组蛋白上的酶。
表1. 组蛋白磷酸化位点、已知激酶和功能 [2,16]
| 组蛋白 | 磷酸化位点 | 生物体 | 激酶 | 生物学功能 |
|---|---|---|---|---|
| H2A | S1 | 哺乳动物 | MSK | 有丝分裂,抑制转录 |
| T119 | 哺乳动物 | NHK-1, AURKB | 有丝分裂期间染色质结构和功能的调节 | |
| S121 | 裂殖酵母 | Bub1 | 通过在姐妹着丝粒处招募舒格神蛋白来维持染色体稳态 | |
| S129 | 酵母 | Mec1, Tel1 | DNA双链断裂修复 | |
| S139 (H2AX) | 哺乳动物 | ATR, ATM, DNA-PKcs, RSK2, MSK1 | DNA修复,减少EGF介导的细胞转化,凋亡 | |
| Y142 (H2AX) | 哺乳动物 | WSTF | 细胞存活和凋亡之间的决定 | |
| H2B | S10 | 酵母 | Ste20 | 凋亡 |
| S14 | 各种脊椎动物 | MstI | 凋亡 | |
| S32 | 哺乳动物 | PKC | EGF信号传导 | |
| S33 | 果蝇 | CTK-TAF1 | 转录调控 | |
| S36 | 哺乳动物 | AMPK | 直接的转录和染色质调控途径,引导细胞对应激的响应 | |
| H3 | T3 | 哺乳动物 | Haspin | 有丝分裂 |
| T6 | 哺乳动物 | PKCβ | 雄激素依赖性H3T6磷酸化阻止LSD1介导的H3K4去甲基化,维持激素依赖性基因的活化 | |
| S10 | 酵母, 哺乳动物 | Snf1, IpL1 (酵母), AURKB (哺乳动物), MSK1, MSK2, IKKα, PKB/Akt, Rsk2, PIM1 | 转录,染色质浓缩,UVB应答 | |
| T11 | 哺乳动物 | Chk1, PRK1, Dlk/Zip kinase | 减数分裂,转录,DNA损伤应答 | |
| S28 | 哺乳动物 | AURKB, ERK1/ERK2, p38 | 减数分裂 | |
| MLTK-α, JNK1/2, MSK1 | 有丝分裂,转录 | |||
| Y41 | 哺乳动物 | JAK2 | 转录 | |
| T45 | 哺乳动物, 芽殖酵母 | PK-Cδ, Cdc7-Dbf4 | 凋亡,在DNA损伤时,当DNA被剪裂时的作用,DNA复制 | |
| H4 | S1 | 酵母, 哺乳动物 | CKII | DNA修复,转录 |
| Sps1 | 减数分裂和染色质装配,转录 | |||
| S47 | 酵母, 哺乳动物 | PAK2 | (H3.3-H4)沉积 | |
| H18/75 | 哺乳动物 | - | 通过破坏组蛋白八聚体来促进DNA复制的进行 | |
| H1 | S/T | CDK2 | 有丝分裂,转录 |
2.2 擦除酶
组蛋白磷酸酸酶,也被称为擦除酶,从磷酸化组蛋白上去除磷酸基团。在哺乳动物中有八种不同的Ser/Thr特异性蛋白磷酸酶(PPs),包括PP1、PP2A、PP2B、PP2C、PP4、PP5、PP6和PP7 [3]。在这些酶中,PP1、PP2A和PP4都被确认为组蛋白磷酸酸酶。
| 组蛋白磷酸激酶 | 识别去磷酸化 | 生物体 |
|---|---|---|
| PP1 | H1 [4] | 细胞周期依赖性 |
| PP1 homologue | H3S10 [5] | 酵母和蠕虫,在有丝分裂期间 |
| PP2A | H3S10ph [6] | 果蝇 |
| γ-H2AX [7] | 哺乳动物和酵母 | |
| PP4 | γ-H2AX [8] | 哺乳动物和酵母 |
2.3 读取蛋白
组蛋白磷酸化的读取蛋白是识别并结合磷酸化组蛋白的磷酸化组蛋白结合蛋白,从而将信号解释为不同的生物学输出。这些蛋白携带磷酸结合结构域,例如14-3-3和BRCT结构域。
3. 组蛋白磷酸化机制
了解组蛋白磷酸化需要掌握其潜在机制:
3.1 激酶激活
组蛋白磷酸化始于激酶的激活。这些激酶可以在细胞应激、生长因子和DNA损伤等各种信号作用下被激活。
3.2 磷酸添加
一旦激活,激酶会靶向组蛋白蛋白质上特定的氨基酸,主要是丝氨酸、苏氨酸和酪氨酸残基。磷酸基团被添加到这些氨基酸上,改变了组蛋白的电荷和结构。
3.3 功能结果
组蛋白磷酸化通过多种机制影响染色质结构和基因表达。它可以创造其他蛋白质结合位点,或者直接改变组蛋白和DNA之间的相互作用。这些变化最终决定了附近基因是被激活还是被沉默。
4. 组蛋白磷酸化的功能
由激酶和磷酸酶协调的组蛋白磷酸化和去磷酸化被认识为调控各种涉及染色质的过程的关键机制,包括转录、DNA复制、DNA损伤修复、染色体分离和凋亡反应。
例如,H2A.X S139 和 H4S1 与 DNA 损伤应答相关。H3S10、H3S28、H2BS32、H3T6、H3T11、H3Y41、H2BS36、H2BY37、H4S1 和 H4S47 与转录相关。H3S10 和 H3S28 出现在有丝分裂中。H4S1 和 H2BS10 见于减数分裂。H2BS10、H2AXS139 和 H3T45 参与了凋亡。
组蛋白磷酸化还在细胞周期调控中发挥着重要作用。在G1期,组蛋白磷酸化促进细胞生长和DNA复制。在S期,组蛋白磷酸化有助于维持复制叉的稳定性。在G2和M期,组蛋白磷酸化对染色体分离和有丝分裂过程至关重要。
对于变异型组蛋白H2A(X)在丝氨酸139位置的磷酸化,即γH2A(X),是DNA损伤应答(DDR)中的关键事件。这种修饰在细胞周期的各个阶段以及对各种DDR途径的应答中发生,包括非同源末端连接(NHEJ)、同源重组(HR)和与复制有关的DNA修复。
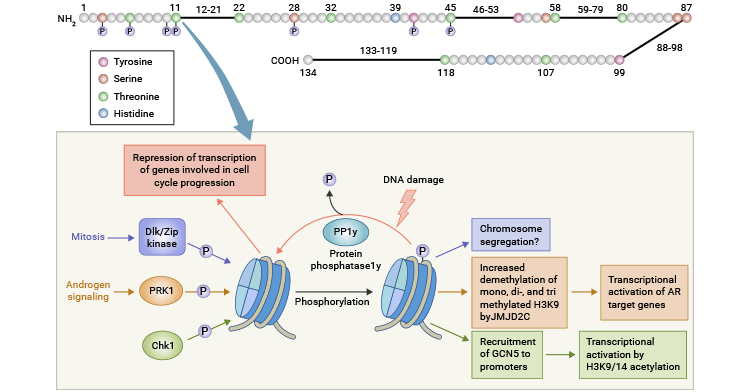
图1. 哺乳动物细胞中组蛋白H2AX磷酸化位点和发生在S139的重要事件 [16]
5. 组蛋白磷酸化与其他组蛋白修饰的相互作用
组蛋白磷酸化,类似于其他组蛋白修饰,不是独立发挥作用的,而是与其他翻译后修饰,包括甲基化和乙酰化,进行复杂的相互作用。
H3S10磷酸化在物理上和功能上与几乎相邻的K14残基的乙酰化相结合,而H3K14的乙酰化增强了H3S10ph与14-3-3之间的相互作用。Zippo等人发现,H3S10ph能与14-3-3蛋白相互作用,并促使组蛋白乙酰转移酶MOF的招募,MOF乙酰化H3K16。这些连续事件招募了Brd4和P-TEFb,最终促进了局部转录 [13]。
H3S28ph与H3K27ac一起有助于启动转录。它通过将染色质中的Polycomb抑制复合物排斥,启动附近H3K27残基的去甲基化和随后的乙酰化,最终导致转录的激活。
PKCβI介导的H3T6的磷酸化抑制了LSD1和JARID1B对H3K4的去甲基化,而PRK1介导的H3T11的磷酸化促进了JMJD2C对H3K9的去甲基化 [14, 15]。
一项研究表明,H3T11磷酸化增加了它在Gcn5依赖的基因启动子上与KAT Gcn5的相互作用,如细胞周期调节因子cyclin B和cdk1,最终导致H3K9和K14的乙酰化增强,从而推动了转录的激活。
H4乙酰转移酶MOF通过与14-3-3的相互作用被招募到H3S10的磷酸化区域,从而在即时早期(IE)基因的激活期间实现H3S10ph和H4K16ac之间的相互作用 [13]。
6. 组蛋白磷酸化与疾病
组蛋白磷酸化与许多疾病的发生和发展密切相关,包括肿瘤、心血管疾病和神经系统疾病。在肿瘤中,异常的组蛋白磷酸化导致基因表达失调,促使异常细胞增殖和转移。多种癌症类型中都有多个组蛋白蛋白的异常磷酸化,以及负责调控组蛋白磷酸化的基因突变。在心血管疾病中,组蛋白磷酸化的不平衡可以导致心肌细胞凋亡和心肌梗死。在神经系统疾病中,组蛋白磷酸化的失调可能导致神经元死亡和认知下降。
参考文献:
[1] Stevely WS, and Stocken LA (1966) Phosphorylation of rat-thymus histone [J]. Biochem. J 100, 20C-21C.
[2] Lau, P.N.I., and Cheung, P. (2009). Histone Phosphorylation: Chromatin Modifications that Link Cell Signaling Pathways to Nuclear Function Regulation [J]. In Handbook of Cell Signaling 2nd edition, R. A. Bradshaw and E. A. Dennis, eds. (Oxford: Academic Press), pp. 2399–2408.
[3] Moorhead GB, Trinkle-Mulcahy L, Ulke-Lemee A (2007). Emerging roles of nuclear protein phosphatases [J]. Nat Rev Mol Cell Biol 8: 234–244.
[4] Paulson JR, Patzlaff JS, Vallis AJ (1996). Evidence that the endogenous histone H1 phosphatase in HeLa mitotic chromosomes is protein phosphatase 1, not protein phosphatase 2A [J]. J Cell Sci 109: 1437–1447.
[5] Hsu JY et al. (2000). Mitotic phosphorylation of histone H3 is governed by Ipl1/aurora kinase and Glc7/PP1 phosphatase in budding yeast and nematodes [J]. Cell 102: 279–291.
[6] Nowak SJ, Pai CY, Corces VG (2003). Protein phosphatase 2A activity affects histone H3 phosphorylation and transcription in Drosophila melanogaster [J]. Mol Cell Biol 23: 6129–6138.
[7] Chowdhury D, Keogh MC, et al. (2005) γ-H2AX dephosphorylation by protein phosphatase 2A facilitates DNA double-strand break repair [J]. Mol Cell 20: 801–809.
[8] Chowdhury D, Xu X, et al. (2008) A PP4-phosphatase complex dephosphorylates γ-H2AX generated during DNA replication [J]. Mol Cell 31: 33–46.
[9] Stucki, M., Clapperton, et al. (2005). MDC1 directly binds phosphorylated histone H2AX to regulate cellular responses to DNA double-strand breaks [J]. Cell 123, 1213–26.
[10] Kelly, A.E., Ghenoiu, C., et al. (2010). Survivin Reads Phosphorylated Histone H3 Threonine 3 to Activate the Mitotic Kinase Aurora B [J]. Science 330, 235–239.
[11] Wang, F., Dai, J., et al. (2010). Histone H3 Thr-3 Phosphorylation by Haspin Positions Aurora B at Centromeres in Mitosis [J]. Science 330, 231–235.
[12] Yamagishi, Y., Honda, T., Tanno, Y., and Watanabe, Y. (2010). Two Histone Marks Establish the Inner Centromere and Chromosome Bi-Orientation [J]. Science 330, 239– 243.
[13] Zippo A, Serafini R, Rocchigiani M et al. (2009) Histone crosstalk between H3S10ph and H4K16ac generates a histone code that mediates transcription elongation [J]. Cell 138:1122–1136.
[14] Metzger, E., Yin, N., et al. (2008). Phosphorylation of histone H3 at threonine 11 establishes a novel chromatin mark for transcriptional regulation [J]. Nature Cell Biology 10, 53–60.
[15] Metzger, E., Imhof, A., et al. (2010). Phosphorylation of histone H3T6 by PKC|[bgr]|I controls demethylation at histone H3K4 [J]. Nature 464, 792.
[16] Banerjee T, Chakravarti D. A peek into the complex realm of histone phosphorylation. Molecular and Cellular Biology [J]. 2011 Dec;31(24):4858-4873.






