组蛋白修饰和DNA甲基化
表观遗传变化涵盖了DNA结构的改变,这些改变源于DNA的复制后修饰和DNA相关蛋白的翻译后修饰。与突变不同,表观遗传变化迅速发生且可逆。在这些变化中,DNA甲基化和组蛋白蛋白质修饰是显著的表观遗传机制,它们错综复杂地相互联系。
1. 组蛋白修饰
组蛋白修饰是指发生在组蛋白蛋白质上的翻译后化学修饰,包括乙酰化、甲基化、磷酸化、泛素化、ADP-核糖基化和SUMO化等,它们在细胞核内调控染色质结构和基因表达中起着至关重要的作用。这些修饰可以通过改变染色质的结构来促进或抑制基因表达,并且对于各种细胞过程至关重要,包括转录调控、DNA修复和表观遗传。
2. DNA甲基化
DNA甲基化是由DNA甲基转移酶(DNMT)催化的一种表观遗传修饰,它将一个活性甲基基团从S-腺苷甲硫氨酸(SAM)转移到胞嘧啶的嘧啶环的第五位置,形成5-甲基胞嘧啶(5meC)[5]。DNA甲基化改变染色体结构、DNA构象和DNA稳定性,从而通过招募负责基因抑制的蛋白质或阻碍转录因子与DNA之间的相互作用来调节基因表达。
| 参与DNA甲基化的酶或蛋白 | 功能 | ||
|---|---|---|---|
| Writers | Dnmt1, Dnmt3a, Dnmt3b, Dnmt3L | 催化将甲基基团添加到胞嘧啶残基上 | |
| Erasers | AID/APOBEC, TDG, SMUG1 | 修饰和去除甲基基团 | |
| Readers | MBD proteins | MeCP2, MBD1, MBD2, MBD3, MBD4 | 识别并结合甲基基团,最终影响基因表达 |
| UHRF proteins | UHRF1, UHRF2 | ||
| Zinc-finger proteins | Kaiso, ZBTB4, ZBTB38 | ||
在哺乳动物中,DNA甲基化发生在基因组中的各种背景下 [6]。然而,在体细胞中,超过98%的DNA甲基化主要发生在胞嘧啶-鸟嘌呤序列(CpG)二核苷酸序列内,而在胚胎干细胞(ESCs)中,高达25%的甲基化事件发生在非CpG背景下 [6]。CpG岛是相对富含CpG序列的区域,通常由于受到保护而处于非甲基化状态(除了位于失活的X染色体上和印迹基因上的基因)。
DNA甲基化通常在合子形成期间被抹去,并在着床周围的胚胎中重新建立 [7]。大部分DNA甲基化对于正常发育至关重要,并在各种重要过程中发挥关键作用,例如基因组印记、X染色体失活,以及抑制转录和转座子元件的转录 [5, 8]。DNA甲基化的失调可能导致癌症等疾病。
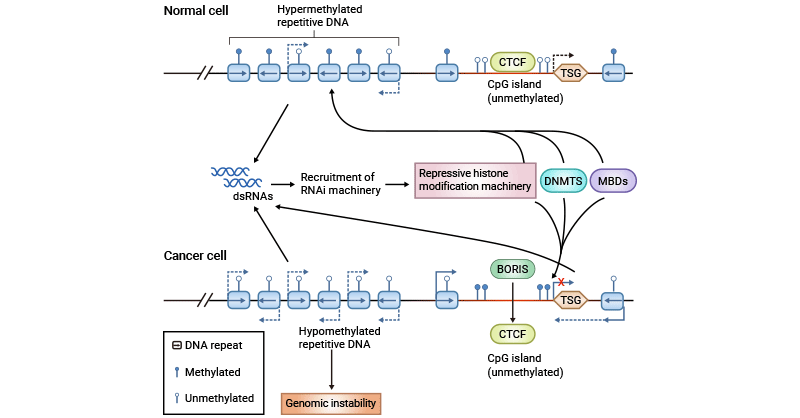
图1. DNA甲基化和癌症
图片参考来源: https://www.nature.com/articles/nrg1655
3. 组蛋白修饰和DNA甲基化的关联
染色质中的DNA甲基化不是独立进行的。相反,DNA甲基化与多种组蛋白修饰之间存在错综复杂的相互作用,包括乙酰化、甲基化和泛素化。
初步研究表明,DNA甲基化与组蛋白修饰之间的联系是通过两类甲基化CpG DNA结合蛋白介导的:MBD家族(MeCP2和MBD1-4)和BTB/POZ家族(Kaiso/ZBTB 33和ZBTB4/38)[1]。这些甲基化CpG结合蛋白不仅与甲基化的DNA结合,还与多个不同的染色质修饰酶结合,包括组蛋白去乙酰化酶(HDACs)和组蛋白甲基转移酶,从而介导DNA甲基化和组蛋白修饰之间的相互作用,形成抑制性的染色质结构 [2-4]。
3.1 组蛋白甲基化和DNA甲基化
DNA甲基化可以通过涉及DNMTs、H3K9甲基转移酶SUV39H1/2和甲基化CpG结合结构域蛋白的相互作用引导H3K9甲基化 [9]。此外,H3K27的三甲基化与底层DNA甲基化紧密相连,其机制涉及到H3K27甲基转移酶EZH2与DNA甲基转移酶的直接相互作用 [10]。
组蛋白修饰也可以影响DNA甲基化模式。例如,Dnmt3L与H3组蛋白尾部结合并招募Dnmt3a和Dnmt3b来启动DNA甲基化 [11]。此外,Dnmt3a与H3组蛋白尾部的直接相互作用,有时候会被抑制性组蛋白标记H3K36m3所促进,从而增强其甲基转移酶活性 [12]。然而,活性组蛋白修饰H3K4me3的存在会破坏Dnmt3a、Dnmt3b和Dnmt3L与H3组蛋白尾部的结合,阻止甲基化的进行 [11]。
3.2 组蛋白乙酰化和DNA甲基化
MBD蛋白与组蛋白去乙酰化酶(HDAC)酶相互作用,通过从组蛋白的赖氨酸残基中去除乙酰基,促使转录抑制性染色质环境的形成 [13]。
Dnmt1和Dnmt3b均可以与从组蛋白上去除乙酰基的HDACs相互作用,导致DNA的紧缩,限制了转录的访问 [14]。
研究还表明,增强的组蛋白乙酰化可以诱导DNA去甲基化 [15]。
3.3 组蛋白泛素化和DNA甲基化
Atsuya Nishiyama及其同事证明,Uhrf1介导的组蛋白H3泛素化是维持DNA甲基化的关键先决条件 [16]。Uhrf1对半甲基化DNA具有特殊亲和力,这是由其SRA(SET和RING指环
指)结构域促成的,并且在维持DNA甲基化方面发挥了关键作用,通过将Dnmt1招募到半甲基化DNA位点。
Luna Yamaguchi等人表明,Usp7参与维持DNA甲基化的调控,它通过去泛素化Uhrf1介导的组蛋白H3泛素化来发挥作用 [17]。Jialun Li等人发现,USP7在负调控整体DNA甲基化,并通过减轻依赖于组蛋白泛素化的DNMT1的招募来保护基因组免受过度DNA甲基化的伤害 [18]。
组蛋白修饰和DNA甲基化在表观遗传调控中紧密相连。这些过程共同协作,编织出基因活性或沉默的动态表观遗传景观,对发育、疾病和细胞身份的各种生物学背景都起着至关重要的作用。了解组蛋白修饰和DNA甲基化之间的相互作用对于揭示复杂的基因调控网络及其在各种生物学背景中的影响至关重要。
参考文献:
[1] O. Bogdanovic, G.J. Veenstra. DNA methylation and methyl-CpG binding proteins: developmental requirements and function [J]. Chromosoma, 118 (2009), pp. 549-565.
[2] Nan X, Ng HH, Johnson CA, Laherty CD, Turner BM, Eisenman RN, et al. Transcriptional repression by the methyl-CpG-binding protein MeCP2 involves a histone deacetylase complex [J]. Nature. 1998;393:386–389.
[3] Hendrich B, Bird A. Identification and characterization of a family of mammalian methyl-CpG binding proteins [J]. Mol Cell Biol. 1998;18:6538–6547.
[4] Bird A. DNA methylation patterns and epigenetic memory [J]. Genes Dev. 2002;16:6–21.
[5] Robertson KD. DNA methylation and human disease [J]. Nat Rev Genet. 2005;6(8):597-610.
[6] Lister R, Pelizzola M, Dowen RH, et al. Human DNA methylomes at base resolution show widespread epigenomic differences [J]. Nature. 2009; 462(7271):315-22v.
[7] Zhu JK. Active DNA demethylation mediated by DNA glycosylases [J]. Annu Rev Genet. 2009;43:143-66.
[8] Gopalakrishnan S, Van Emburgh BO, Robertson KD. DNA methylation in development and human disease [J]. Mutat Res. 2008;647(1-2):30-8.
[9] Lehnertz B, Ueda Y, Derijck AA, et al. Suv39h-mediated histone H3 lysine 9 methylation directs DNA methylation to major satellite repeats at pericentric heterochromatin [J]. Curr Biol. 2003;13(14):1192-200.
[10] Vire E, Brenner C, Deplus R, et al. The polycomb group protein EZH2 directly controls DNA methylation [J]. Nature. 2006;439(7078):871-4.
[11] Ooi SK, Qiu C, et al. (2007) DNMT3L connects unmethylated lysine 4 of histone H3 to de novo methylation of DNA [J]. Nature 448: 714–717.
[12] Dhayalan A, Rajavelu A, et al. (2010) The Dnmt3a PWWP domain reads histone 3 lysine 36 trimethylation and guides DNA methylation [J]. J Biol Chem 285: 26114–26120.
[13] Ng HH, Zhang Y, et al. 1999. MBD2 is a transcriptional repressor belonging to the MeCP1 histone deacetylase complex [J]. Nat Genet 23: 58–61.
[14] Geiman TM, Sankpal UT, et al. (2004) DNMT3B interacts with hSNF2H chromatin remodeling enzyme, HDACs 1 and 2, and components of the histone methylation system [J]. Biochem Biophys Res Commun 318: 544–555.
[15] Cervoni N, Szyf M (2001). Demethylase activity is directed by histone acetylation [J]. J Biol Chem 276: 40778–40787.
[16] Nishiyama, A. et al. Uhrf1-dependent H3K23 ubiquitylation couples maintenance DNA methylation and replication [J]. Nature 502, 249–253 (2013).
[17] Yamaguchi, L., Nishiyama, A., Misaki, T. et al. Usp7-dependent histone H3 deubiquitylation regulates maintenance of DNA methylation [J]. Sci Rep 7, 55 (2017).
[18] Li, J., Wang, R., Jin, J. et al. USP7 negatively controls global DNA methylation by attenuating ubiquitinated histone-dependent DNMT1 recruitment [J]. Cell Discov 6, 58 (2020).






