DNA损伤与修复
日期:2024-01-24 13:10:15
DNA是遗传信息的宝库,是生物体生存和繁殖的必要条件,因此保持 DNA分子的完整性对细胞至关重要。然而,DNA在不断复制并传递给子细胞的过程中难免会受到损伤。那么,细胞如何处理受损的 DNA呢?到底什么是 DNA损伤?DNA损伤的原因是什么?DNA损伤与哪些疾病相关?
本文将介绍DNA损伤及其原因,与突变的区别,以及DNA损伤的类型、DNA损伤反应途径和相关疾病。
1. 什么是DNA损伤?
DNA损伤是指任何偏离其原始双螺旋结构的改变。所有细胞生物都会发生DNA损伤。
2. 什么导致DNA损伤?
DNA损伤的来源很多,主要可分为两类:内源性因素和外源性因素。细胞内源性DNA损伤包括复制错误和内源性化学反应引起的自发性基因毒性损伤。外源又可分为物理因素和化学因素。
3. DNA损伤的类型
每个细胞在其生命周期中都必须不断对抗内源性和外源性DNA损伤 [1]。
3.1 内源性DNA损伤
据估计,每个细胞每天要经历近10万次自发的DNA损伤 [2]。
3.1.1 DNA复制错误
以DNA为模板进行碱基配对的DNA复制是一项严格而精确的工作,但并非完全没有错误。碱基配对错误的频率约为10-1-10-2。在DNA复制酶的作用下,碱基配对错误的频率降低到约10-5-10-6。如果复制过程中出现错误的核苷酸,DNA聚合酶也会暂停催化。虽然复制过程中碱基错配的概率很低,但仍有一些错配碱基逃过了校对甚至是MMR机制的检测。当模板受损时,反式损伤合成(TLS)聚合酶的保真度很低,是自发突变的重要来源。此外,拓扑异构酶、尿嘧啶掺入等也会造成错误。
虽然大多数DNA复制的保真度相当高,但有时也会出现错误。核苷酸碱基的插入和缺失都有可能发生。此外,DNA复制过程中的自发错误可能会导致在新生合成的DNA分子中加入错误的核苷酸,造成碱基对不匹配 [3]。
3.1.2 水解DNA损伤
DNA的水解损伤包括单个碱基的脱氨和缺失。各种代谢产物的生化反应和过量的活性氧(ROS)都可能导致水解损伤。
● 碱基脱氨
含氨基的含氮碱基会自发脱氨基,形成C-U、A-I(次黄嘌呤和G-X(黄嘌呤)。有趣的是,单链中碱基脱氨基的频率远高于双链,因此复制、转录和重组过程中的瞬时单链状态会加剧这种损伤,并在修复突变效应之前发生。在所有碱基中,5-甲基胞嘧啶发生脱氨的频率最高,生成的G:T碱基对会通过较慢的错配修复(MMR)得到纠正。分散的CpG序列很容易受到胞嘧啶-5-甲基转移酶的影响。脊椎动物约有70%-80%的CpG胞嘧啶被甲基化。由其脱氨引起的GC→AT碱基转换占人类遗传病点突变的三分之一。
● 碱基缺失
DNA碱基缺失又称AP(嘌呤/近嘧啶)位点,其突变性特别强,如果不及时修复,会抑制转录。碱基也会丢失。大肠杆菌每代大约丢失一个嘌呤,而哺乳动物细胞每天大约丢失 10,000 个嘌呤。相对而言,嘧啶的 N-糖苷键相对稳定,丢失的概率仅为嘌呤的1/20。消旋位点不稳定,容易发生β-消除反应,导致单链断裂(SSB)。
● 碱基同分异构
DNA中四种碱基的同分异构体均可自发发生变化(例如,烯醇碱基和酮碱基之间的相互转化)。这种变化会改变碱基配对之间的氢键。腺嘌呤可以与胞嘧啶配对,胸腺嘧啶可以与鸟嘌呤配对,等等。如果这些配对在DNA复制过程中发生,就会造成子代DNA序列与亲代DNA序列不同的错误损伤。碱基具有酮和烯醇结构的自发同素异形作用,会造成碱基错配。例如,当腺嘌呤形成A=NH结构时,可形成A=C配对;当鸟嘌呤形成G-OH结构时,可形成GT三键配对。
3.1.3 细胞代谢的内源性副产物
活性氧(ROS)也会造成DNA损伤。活性氧自由基可攻击碱基上的双键,引起开环反应等,还可破坏核糖磷酸骨架,造成单链断裂。这与电离辐射类似。据估计,活性氧在哺乳动物细胞中每小时可造成2300次单链断裂。产生ROS的DNA损伤是最常见的损伤。
3.2 外源性DNA损伤
环境DNA损伤可由物理或化学来源产生。
3.2.1 物理因素
常见的物理因素主要包括电离辐射(IR)和太阳紫外线(UV)。
射线、接触放射性物质和使用放射疗法进行的医疗。它可以诱导碱基修饰、链间交联和DNA链断裂,尤其是双链断裂(DSB)。此外,红外线还会诱导形成ROS,从而进一步损伤 DNA。
来自太阳的紫外线会与DNA发生反应,主要导致两个相邻的嘧啶形成二聚体,阻碍DNA复制和转录。
红外线包括X射线、伽马射线、α和β粒子以及中子。日常生活中的红外线通常来自宇宙辐射和使用X射线或放射治疗的医疗手段。它通过产生DNA断裂,尤其是双链断裂(DSB),直接影响DNA结构。红外线还会导致活性氧(ROS)的形成,从而产生嘌呤/近嘧啶(abasic)位点、单链断裂(SSB)、糖分子修饰和脱氨基加成碱基 [4] [5]。
3.2.2 化学因素
导致DNA损伤的化学事件包括水解、接触活性氧物质(ROS)和其他活性代谢物。
作为相对常见的代谢副产物,ROS可导致单链和双链断裂、加成和交联。
导致DNA损伤的化学因素有很多。烷化剂具有活性烷基,可转移到碱基或磷酸上,如硫酸二甲酯、甲磺酸甲酯(MMS)、芥子气等。鸟嘌呤的O6和N7最容易被烷基化,导致错配(GT)或脱落。磷酸三酯不稳定,容易断裂。双官能烷基化剂可导致交联,被称为交联剂。某些烷化剂如环磷酰胺(cyclophosphamide)可用于化疗。碱基或核苷类似物,如5-氟尿嘧啶(FU)、5-溴尿嘧啶(BrdU)、6-巯基嘌呤等,可竞争性抑制核苷酸合成或结合核酸造成错配。亚硝酸盐可导致碱基脱氨,亚硝胺被氧化后产生烷化剂和自由基。还有一类化学物质叫代谢活化化合物,在肝脏混合功能氧化酶(细胞色素P450)的催化下,形成活性烷化剂或环氧化物等,与核酸相互作用,引起突变。如芳香胺、多环芳烃等。苯并芘是致癌性最强的多环芳烃之一。烟草烟雾中含有大量的芳香胺和多环芳烃。黄曲霉毒素也是一种代谢活化致癌物,其中黄曲霉毒素B1的作用最强。黄曲霉毒素B1被动扩散到细胞内后,经P-450复合物代谢为活性形式的黄曲霉毒素B1-8,9-环氧化物,然后加入鸟嘌呤N7,使糖苷键减弱,导致脱嘌呤。
4. DNA损伤与DNA变异
虽然DNA损伤和DNA突变都是DNA错误的一种,但它们之间有很大的不同。DNA损伤是DNA的物理异常,如单链和双链断裂,而突变则是DNA碱基序列的改变。DNA损伤可以被酶识别,因此,如果有多余的信息可供复制,就可以正确修复。虽然大多数DNA损伤可以进行DNA修复,但未修复的DNA损伤会在复制细胞中积累,从而导致突变。与DNA损伤不同,DNA突变一旦在两条DNA链上都出现碱基变化,酶就无法识别和修复,从而导致蛋白质功能和调节发生改变。
DNA损伤和DNA变异的主要区别在于,DNA损伤是DNA结构的改变,而DNA变异是DNA核苷酸序列的改变。此外,DNA损伤会阻止DNA复制,而突变则会改变DNA编码的遗传信息。DNA损伤和突变是DNA中发生的一组错误。此外,环境因素以及新陈代谢释放的化合物都会造成DNA损伤,而突变主要是由于DNA复制和重组过程中的错误造成的。
5. DNA损伤应答信号通路
所有活细胞都不可避免地会发生不同类型和程度的DNA损伤。为了在这些不利条件下存活下来,并将完整的遗传信息忠实地传递给下一代,细胞进化出了一种高度有序和协调的机制,即DNA损伤应答(DDR),以减轻这些基因毒性应激。
DDR是维持基因组完整性和稳定性的关键因素。DDR传感蛋白可识别受损DNA并启动一连串信号转导因子(如 ATM、ATR 或 Rad17-RFC 复合物),从而激活DDR介导因子(如丝氨酸/苏氨酸激酶 Chk1、Chk2、Cdc25 磷酸酶)。活化的 DDR介质可激活特定的蛋白质机制,触发适当的细胞反应,可能是DNA修复、细胞周期检查点激活、诱导细胞凋亡或衰老或病变耐受 [6-9]。这种不同的反应取决于细胞类型、损伤程度和其他因素。
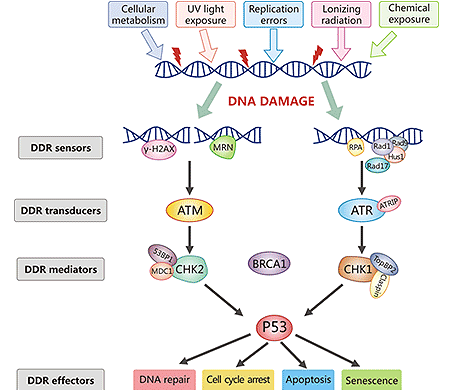
图1. DNA损伤应答信号通路
图片来源:https://www.sciencedirect.com/science/article/abs/pii/S156878641830096X
5.1 DNA修复机制
DNA损伤修复系统的激活是DNA损伤应答的主要终点。真核细胞表现出多种DNA修复机制来处理潜在的DNA损伤 [10]。这些修复途径的保真度和诱变后果各不相同 [10]。这些机制可感知和识别所有类型的DNA损伤,暂停基因组复制,发出修复信号,并纠正或容忍DNA病变 [11]。不同修复机制之间几乎没有冗余。
DNA修复机制包括直接修复、碱基切除修复、核苷酸切除修复、双链断裂修复和交联修复。
5.1.1 切除修复机制
切除修复机制的目标是去除大块DNA加合物和紫外线诱导的光产物、碱基对改变和嘌呤丢失、DNA错配以及单链和双链DNA断裂。
● 核苷酸切除修复(NER)
核苷酸切除修复(NER)是用途最广的DNA修复途径,可修复多种物理和化学因素导致的DNA损伤,如紫外线辐射介导的和致癌的DNA化学加合物。NER可以切除DNA损伤的大片段。它可以修复DNA损伤的各种形式,包括6-4光产物、环丁烷嘧啶二聚体和大块加合物。
● 碱基切除修复(BER)
碱基切除修复(BER)是一种特别有助于清除小碱基修饰的机制。它可以修复氧化、脱氨基、烷基化和消旋单碱基损伤,这些损伤一般不会导致DNA螺旋发生明显扭曲。在BER途径中,DNA糖基化酶检测并消除受损碱基,形成位点,然后由AP内切酶切割,留下一个碱基间隙。DNA聚合酶和DNA连接酶进一步固定该间隙。
5.1.2 错配修复(MMR)
MMR途径可纠正碱基误配,包括碱基错配、插入和缺失。该修复系统有助于识别DNA螺旋扭曲、区分两条链并消除复制错误。通过纠正躲过复制聚合酶校对的不常见错误,它可将复制保真度提高100倍以上 [12-14]。
5.1.3 DNA双链断裂修复(DSBR)途径
DNA DSB是最具细胞毒性的DNA损伤。DNA DSB修复途径包括同源重组(HR)、DNA末端连接和非同源末端连接(NHEJ)。
● 同源重组(HR)
同源重组是一种精确的修复途径,需要同源DNA序列作为修复模板 [15],仅限于细胞周期的S/G2期 [16]。HR是一个缓慢、复杂和高度精确的修复过程,涉及大量酶和蛋白质。这一途径既能修复单端和双端断裂,也能修复蛋白质阻断的末端。
● 非同源末端连接(NHEJ)
NHEJ 是一种易出错的修复途径,在大多数哺乳动物细胞中主要用于重新连接DSB。NHEJ的发生偏好于G0、G1和早期S期 [17-19]。它是哺乳动物细胞在DNA复制前抵御DSB的第一道防线。与HR相比,NHEJ进展迅速,而且与模板和细胞周期无关。它通常具有突变性,只能修复双端断裂,不能修复蛋白质阻断末端。NHEJ涉及断裂DNA末端的连接,不需要序列同源性。
替代性末端连接(a-EJ)途径可修复DNA双链断裂(DSB),它是通过末端切除产生3'单链来启动的。
5.2 细胞周期停滞和细胞凋亡
如果受损DNA得到及时完全修复,对细胞的影响几乎微乎其微。未修复的DNA可能编码细胞周期所需的蛋白质,从而导致细胞周期停滞。细胞周期对细胞的生长、增殖和繁殖至关重要。它最终会影响细胞的生长和存活。如果损伤过度,细胞不再消耗能量来有效修复损伤,很可能会发生凋亡或衰老。
6. DNA损伤与疾病
要在细胞中表达功能性信使核糖核酸并最终生成蛋白质,DNA 序列的准确读取是不可或缺的。此外,细胞分裂过程中忠实的DNA复制对于子细胞从母细胞继承完整的遗传物质至关重要。因此,由内在和内部因素引起的细胞DNA的各种改变会产生深远的生物学后果。虽然大多数DNA损伤可以修复,但修复系统的效率并非百分之百。未修复的DNA损伤会改变基因表达、抑制细胞分裂或导致细胞死亡 [20-22]。DNA 损伤在诱变和致癌过程中也扮演着重要角色。Tomasetti C及其同事发现,在17种癌症类型中发现的突变中有三分之二是DNA复制错误造成的 [23]。
有证据表明,核DNA(编码大多数细胞RNA和蛋白质)和线粒体DNA的损伤与衰老有关 [24]。
细胞DNA损伤也被证明与许多人类疾病的病因和发展有关,如色素性角化症、共济失调性脊髓侧索硬化症、布卢姆综合征和沃纳综合征。
当然,突变并不是永久的坏蛋。试想一下,如果DNA的修复功能足够完善,能够修复所有的DNA损伤,没有突变的产生,那么就不会有基因的改变,也就不会有进化的原材料。根据不同的条件,DNA损伤可以是致病性的,也可以是治疗性的。
DNA 损伤可导致基因改变,如果涉及控制细胞生长的基因,这些突变可导致癌症的发生。DNA的损伤还可能导致细胞死亡,从而给细胞所在的机体带来严重后果,例如,大脑中不可替代的神经元的丧失。受损DNA的积累也被认为是导致衰老的一些原因。
虽然人类基因组DNA的损伤经常发生,但大多数损伤都能通过各种修复机制成功修复。如果DNA病变没有得到及时修复或过于严重而无法修复,就会引发一些信号事件,导致三种不同的细胞命运,包括衰老、凋亡或癌变。
7. 与DNA损伤修复相关的重组蛋白
DNA修复涉及许多途径,其中一些涉及许多蛋白质。为了帮助研究DNA损伤和修复,j9九游会登录入口首页生物提供了一些相关的重组蛋白。
参考文献:
[1] Sancar A., Lindsey-Boltz L.A., et al. Molecular mechanisms of mammalian DNA repair and the DNA damage checkpoints [J]. Annu. Rev. Biochem. 2004; 73: 39-85.
[2] Lindahl T. Instability and decay of the primary structure of DNA [J]. Nature. 1993; 362: 709-715.
[3] Ganai R.A., Johansson E. DNA Replication—A Matter of Fidelity [J]. Mol. Cell. 2016;62:745–755.
[4] Redon CE, Nakamura AJ, et al. Histone gammaH2AX and poly(ADP-ribose) as clinical pharmacodynamic biomarkers [J]. Clin Cancer Res. 2010;16:4532–4542.
[5] Aparicio T, Baer R, Gautier J. DNA double-strand break repair pathway choice and cancer [J]. DNA Repair. 2014;19:169–175.
[6] Zhou BB, Elledge SJ 2000. The DNA damage response: Putting checkpoints in perspective [J]. Nature 408: 433–439.
[7] Matsuoka S, Huang M, Elledge SJ 1998. Linkage of ATM to cell cycle regulation by the Chk2 protein kinase [J]. Science 282: 1893–1897.
[8] Reinhardt HC, Aslanian AS, et al. p53-deficient cells rely on ATM- and ATR-mediated checkpoint signaling through the p38MAPK/MK2 pathway for survival after DNA damage [J]. Cancer Cell 2007, 11: 175–189.
[9] Alexandre Maréchal and Lee Zou. DNA Damage Sensing by the ATM and ATR Kinases [J]. Cold Spring Harb Perspect Biol. 2013 Sep; 5(9): a012716.
[10] Lombard D.B., Chua K.F., et al. DNA repair, genome stability, and aging [J]. Cell. 2005; 120: 497-512.
[11] Hoeijmakers JHJ (2009). DNA damage, aging, and Cancer [J]. New England Journal of Medicine 361:1475–1485.
[12] Kunkel TA. DNA replication fidelity [J]. J Biol Chem. 2004;279(17):16895–16898.
[13] Kunkel TA. Evolving views of DNA replication (in)fidelity [J]. Cold Spring Harb Symp Quant Biol. 2009;74:91–101.
[14] Kunkel TA. Balancing eukaryotic replication asymmetry with replication fidelity [J]. Curr Opin Chem Biol. 2011;15(5):620–626.
[15] Ranjha, L., Howard, S. M. & Cejka, P. Main steps in DNA double-strand break repair: an introduction to homologous recombination and related processes [J]. Chromosoma 127, 187–214 (2018).
[16] Lodovichi, S. et al. Effect of BRCA1 missense variants on gene reversion in DNA double-strand break repair mutants and cell cycle-arrested cells of Saccharomyces cerevisiae [J]. Mutagenesis 35, 189–195 (2020).
[17] Roth, D. B. & Wilson, J. H. Relative rates of homologous and nonhomologous recombination in transfected DNA [J]. Proc. Natl Acad. Sci. USA 82, 3355–3359 (1985).
[18] Deckbar D, Jeggo PA, et al. Understanding the limitations of radiation-induced cell cycle checkpoints [J]. Crit Rev Biochem Mol Biol. 2011;46:271–283.
[19] Chang, H. H. Y., Pannunzio, N. R., Adachi, N., and Lieber, M. R. (2017). Non-homologous DNA end joining and alternative pathways to double-strand break repair [J]. Nat. Rev. Mol. Cell Biol. 18, 495–506.
[20] Zhou BBS, Elledge SJ. The DNA damage response: putting checkpoints in perspective [J]. Nature. 2000;408:433–439.
[21] Norbury CJ, Hickson ID. Cellular responses to DNA damage [J]. Ann Rev Pharmacol Toxicol. 2001;41:367–401.
[22] Kent S. Gates. An Overview of Chemical Processes That Damage Cellular DNA: Spontaneous Hydrolysis, Alkylation, and Reactions with Radicals [J]. Chem Res Toxicol. 2009 Nov; 22(11): 1747–1760.
[23] Tomasetti C, Li L, Vogelstein B. Stem cell divisions, somatic mutations, cancer etiology, and cancer prevention [J]. Science. 2017;355(6331):1330–4.
[24] Karanjawala Z.E., Lieber M.R. DNA damage and aging [J]. Mech. Ageing Dev. 2004; 125: 405-416.











