组蛋白乙酰化
1964年,文森特·奥尔弗里及其团队首次报告了组蛋白乙酰化 [1]。组蛋白乙酰化是一种被广泛研究的组蛋白后转录修饰(PTM),在调控染色质基因转录中发挥着核心作用。
1. 什么是组蛋白乙酰化?
组蛋白乙酰化是指将乙酰基添加到组蛋白蛋白质的N-末端赖氨酸残基上的过程 [1]。所添加的乙酰基的负电荷中和了赖氨酸的正电荷,导致组蛋白与DNA的相互作用减弱,从而使染色质结构更加松弛,有助于转录因子接近基因组序列,从而增加基因表达。
已经检测到各种组蛋白蛋白质上的组蛋白乙酰化。具体而言,H3上的赖氨酸残基K4、K9、K14、K18、K23、K27、K36和K56显示出乙酰化,而H4则在K5、K8、K12、K16、K20和K91上显示出乙酰化。此外,H2A和H2B也经历赖氨酸乙酰化,在H2A上的K5和K9被修饰,在H2B上的K5、K12、K15、K16、K20和K120被乙酰化。
2. 参与组蛋白乙酰化的酶或蛋白
组蛋白乙酰化是一个动态可逆的过程,由组蛋白乙酰化调节因子(HAMs)协同调节,包括“写入者”(组蛋白乙酰转移酶)、“阅读器”和“擦除者”(组蛋白去乙酰化酶),它们分别负责添加、识别和去除乙酰基。HATs和HDACs之间的这种动态相互作用调节了组蛋白乙酰化的平衡,确保了基因表达模式的精确控制。
2.1 组蛋白乙酰转移酶(HATs)
HATs,也称为“写入者”,负责将乙酰基(-COCH3)从乙酰辅酶转移到H3或H4的靶赖氨酸残基的ε-氨基上 [2]。HATS还乙酰化各种非组蛋白底物,因此它们通常被归类为赖氨酸乙酰转移酶(KATs)。
基于它们的细胞定位,HAT可分为两大类:A型HATs位于细胞核,乙酰化核小体组蛋白和其他染色质相关蛋白,而B型HATs位于细胞质,专门乙酰化新合成的游离组蛋白,对转录没有直接影响。
| 组蛋白乙酰转移酶家族 | 成员 | 定位 | 组蛋白亚基 | 功能 | ||
|---|---|---|---|---|---|---|
| Type A HATs | GNAT | KAT2A (GCN5) | Nucleus | H3K9, H3K14, H3K18, H2B | Transcription activation | |
| KAT2B (PCAF) | Nucleus | H3K9, H3K14, H3K18, H2B | Transcription activation | |||
| MYST | KAT5 (Tip60) | Nucleus | H4K5, H4K8, H4K12, H4K16 | Transcription activation, DNA repair | ||
| KAT6A (MOZ/MYST3) | Nucleus | histone H2B, H3K14 and H4K5, H4K8, H4K12 and H4K16 in vitro and H3K9 in vivo [3] | Transcription activation | |||
| KAT6B (MORF/MYST4) | Nucleus | particularly H3 and H4, and nucleosomal histones with a preference for H4 | Transcription activation | |||
| KAT7 (HBO1/MYST2) | Nucleus | H3K14, H4K5, H4K8, H4K12 | Transcription, DNA replication | |||
| KAT8 (MOF/MYST1) | Nucleus | specifically H4K16 | Chromatin boundaries, dosage compensation, DNA repair | |||
| P300/CBP | KAT3B (P300) | Nucleus | all four histone subtypes: H2AK5, H2B (K5, K12, K15, K20), H3 (K14, K18, K27), and H4 (K8, K12) | Transcription activation | ||
| KAT3A (CBP) | Nucleus | |||||
| Others | Transcription co-activators | KAT4 ( TAF1/TBP) | Nucleus | H3 and H4, H3K14 | Transcription activation | |
| KAT12 (TIFIIIC90) | Nucleus | H3K9, H3K14, H3K18 | Transcription activation | |||
| Steroid receptor co-activators | ATAT1 | Nucleus | - | - | ||
| KAT13A (SRC1) | Nucleus | H3, H4 | Transcription activation | |||
| KAT13B (SCR3/AIB1/ ACTR) | Nucleus | |||||
| KAT13C ( p160) | Nucleus | |||||
| KAT13D (CLOCK) | Nucleus | |||||
| Type B HATS | KAT1 (HAT1) | Cytoplasm | H3, H4, H2A | Histone deposition, DNA repair | ||
| HAT4 (NAA60) | Cytoplasm | H4, H2A | - | |||
表格参考来源:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4881052/
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6311713/
2.2 组蛋白去乙酰化酶(HDACs)
HDACs,也被称为“擦除者”,是一种能够从乙酰化的组蛋白中去除乙酰基的酶。组蛋白去乙酰化通过暴露赖氨酸残基的正电荷,使DNA与组蛋白尾部之间发生相互作用,从而减少了DNA的可及性,导致染色质紧缩和更加致密的染色质结构,有效地抑制了基因表达。
HDACs在各种生理过程中至关重要,包括发育和细胞内稳态,同时也参与了神经退行性疾病、遗传疾病和癌症等病理条件的发生。
在人体中,根据它们与酵母的序列同源性,HDAC酶分为四个类别 [4]。I类HDACs包括HDAC1-3和HDAC8。II类HDACs包括HDAC4-7、HDAC9和HDAC10 [5-7]。III类HDACs,也被称为sirtuins,包括SIRT1-7 [8,9],这些酶在活性时需要NAD+辅酶。IV类HDAC只有一种酶HDAC11 [10]。I、II和IV类HDACs是Zn2+依赖的酰胺水解酶。
| 组蛋白去乙酰化酶家族 | 分类 | 亚族 | 成员 | 细胞定位 | 组蛋白亚基 | 生物学功能 |
|---|---|---|---|---|---|---|
| Classical (Zn2+-dependent) | Class I | HDAC1 | Nucleus | all four core histones [11] | Mediates deacetylation of lysine on the N-terminal part of the core histones and plays and important role in transcriptional regulation, cell cycle progression and development event | |
| HDAC2 | Nucleus | all four core histones [11] | ||||
| HDAC3 | Nucleus | H4K5 H4K12 H2AK5 | ||||
| HDAC8 | Nucleus | preferentially deacetylates histones H3 and H4 [12] | ||||
| Class II | Class IIa | HDAC4 | Nucleus | all four core histones [11] | ||
| HDAC5 | Nucleus | all four core histones [11] | ||||
| HDAC7 | Nucleus | |||||
| HDAC9 | Nucleus | H3K9 H3K14 H3K18 [13] |
||||
| Class IIb | HDAC6 | Cytoplasm | all four core histones [11] | |||
| HDAC10 | Cytoplasm | H3K9 H3K14 H3K18 H3K27 [14] |
Involved in MSH2 deacetylation | |||
| Class IV | HDAC11 | Nucleus | H3K9 H3K14 [15] H4K16 H4K5 H4K12 [16] |
Plays an important role in transcriptional regulation, cell cycle progression and developmental events | ||
| NAD+-dependent | Class III | SIRT1 | Nucleus | H3K9 H3K14 H3K56 H4K16 H1K26 [11] |
Chromatin organization, DNA repair/genome stability, cancer | |
| SIRT2 | Cytoplasm | H4K16 H3K56 [11] |
Chromatin condensation/mitosis, DNA repair, cancer | |||
| SIRT3 | Mitochondria | H4K16 [11] | Chromatin silencing, DNA repair, cellular stress | |||
| SIRT4 | Mitochondria | / | Regulates the cellular metabolic response to DNA damage | |||
| SIRT5 | Mitochondria | / | Regulates the mitochondrial lysine succinylome and metabolic networks | |||
| SIRT6 | Nucleus | H3K9 H3K56 [11] |
Telomeric chromatin/senescence, DNA repair/genome stability, energy metabolism | |||
| SIRT7 | Nucleolus | H3K18 [11] | Mediates deacetylation of H3K18ac Mediates deacetylation of lysine residues on the N-terminal part of the core histones |
表格参考来源:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3970420/
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7444376/
2.3 读取蛋白
组蛋白乙酰化读取蛋白特异地识别乙酰化的赖氨酸,并招募转录因子和其他调节蛋白与DNA结合,启动转录,从而激活基因表达 [17,18]。它们通常是含有溴结构域(BRD)的蛋白或者是乙酰化赖氨酸结合蛋白,比如BRD和额外末端结构域(BET)家族,负责将乙酰化信号解释为基因表达和染色质重塑中的特定变化。
BRDs突出显示为唯一能够识别和结合乙酰化组蛋白赖氨酸残基的蛋白群。它们广泛分布在各种组织中,并根据序列或结构相似性被分为八个家族,每个家族都显示出多样的活性,比如组蛋白修饰和染色质重塑。
| BRDs | Members |
|---|---|
| Class I | BAZ1A, BPTF, CECR2, GCN5L2, PCAF |
| Class II | BRD2, BRD3, BRD4, BRDT |
| Class III | BAZ1B, BRD8B, BRWD3, CREBBP, EP300, PHIP, WDR9 |
| Class IV | ATAD2, ATAD2B, BRD1, BRD7, BRD9, BRPF1, BRPF3 |
| Class V | BAZ2A, BAZ2B, SP100, SP110, SP140, SP140L, TRIM24, TRIM33, TRIM66 |
| Class VI | MLL, TRIM28 |
| Class VII | BRWD3, PHIP, TAF1, TAF1L, WDR9, ZMYND8, ZMYND11 |
| Class VIII | ASH1L, PBRM1, SMARCA2, SMARCA4 |
表格参考来源:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7686570/
3. 组蛋白乙酰化的机制
组蛋白酰转移酶(HATs)酰化组蛋白赖氨酸残基,减少了组蛋白和DNA之间的静电相互作用,导致染色质结构松弛。因此,DNA被暴露出来,有助于转录激活因子(TA)被招募到基因组序列上,激活基因表达。
相反,组蛋白去乙酰化酶(HDACs)可以与转录抑制因子(TR)复合物相互作用,去除这些乙酰修饰。这增强了DNA和组蛋白之间的静电相互作用,导致紧凑的染色质,抑制转录。保持组蛋白乙酰化和去乙酰化之间的平衡对于正确的基因表达非常关键。
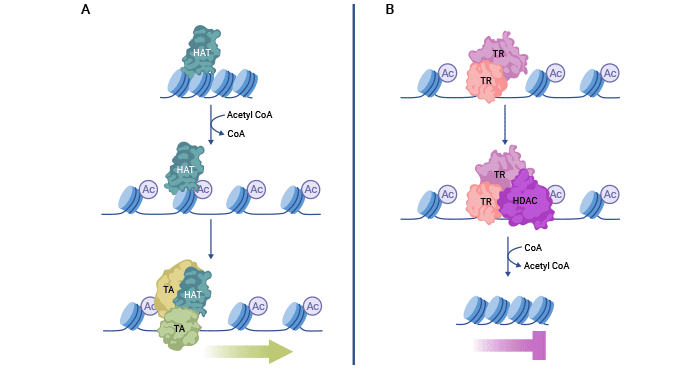
图1. 组蛋白乙酰化与去乙酰化机制
图片参考来源:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7820808/
4. 组蛋白乙酰化的功能
大量研究数据表明,组蛋白乙酰化在细胞中广泛存在,并参与各种细胞活动,包括核小体组装的调节、染色质的折叠和解构、基因转录、DNA损伤修复、细胞周期和凋亡 [19]。
5. 与其他修饰的相互作用
组蛋白乙酰化与其他组蛋白修饰之间存在复杂的相互作用,比如甲基化、磷酸化和泛素化。它们可以相互合作或拮抗,影响染色质结构和基因表达模式。
在酿酒酵母萨克罗米瑟斯小麦,Snf1激酶介导的H3S10ph促进了由Gcn5乙酰转移酶介导的H3K14的乙酰化,在基因激活期间增强了组蛋白H3与14-3-3蛋白Bmh1和Bmh2的相互作用 [20]。在哺乳动物细胞中,H3K18ac和H3K23ac促使CARM1甲基转移酶介导的H3R17甲基化,导致雌激素响应基因的激活 [21]。
组蛋白修饰的相互作用也可以在去除特定修饰方面发挥作用。例如,在酿酒酵母中,与RNA聚合酶II结合的Set2甲基转移酶甲基化H3K36,引导核小体在RNA聚合酶通过后由Rpd3S脱乙酰酶复合物去乙酰化H3和H4 [22]。
Wojcik等人证明,H2A的多个位点,包括K5、K9、K13和K15的乙酰化抑制了H2BK120的泛素化 [23]。
6. 发育和疾病
组蛋白乙酰化参与了许多生物学过程,包括发育、分化和对环境信号的响应。组蛋白乙酰化的失调与各种疾病相关,比如癌症、神经系统障碍和代谢性疾病,突显了它在疾病发病机制中的重要性。
在癌细胞中观察到异常的组蛋白乙酰化模式,导致细胞不受控制地增长和肿瘤发生。HDAC抑制剂能够恢复适当的乙酰化模式,被认为是潜在的治疗某些癌症的方法,提供了一种新的方式来靶向癌细胞而不伤害正常细胞。
在神经退行性疾病中,比如阿尔茨海默病和帕金森病,组蛋白乙酰化的紊乱与与神经细胞存活和功能相关的基因表达模式的变化有关。失调的组蛋白乙酰化可能导致神经退行性病变和疾病进展。
此外,组蛋白乙酰化还与代谢性疾病相关,它可能影响与葡萄糖代谢和脂质稳态有关的基因的表达。对饮食和环境因素的反应引发的表观遗传变化可能导致代谢失调和与肥胖相关的疾病。
总的来说,HATs、HDACs和BRDs形成了一个复杂的调节网络,控制组蛋白的乙酰化状态,从而影响基因对转录的可及性。这种表观遗传机制在各种生物学过程中发挥着关键作用,包括发育、分化和对环境信号的响应。这些酶的失调可能导致基因表达模式的异常,与多种疾病相关,使它们成为治疗干预和药物开发的有希望的靶点。
参考文献:
[1] Allfrey VG, Faulkner R, Mirsky AE. Acetylation and methylation of histones and their possible role in the regulation of RNA synthesis [J]. Proc Natl Acad Sci USA. 64;51:786–794.
[2] Roth, S.Y.; Denu, J.M.; Allis, C.D. Histone acetyltransferases [J]. Annu. Rev. Biochem. 2001, 70, 81–120.
[3] Perez-Campo FM, Costa G, et al. The MYSTerious MOZ, a histone acetyltransferase with a key role in haematopoiesis. Immunology. 2013 Jun;139(2):161-5.
[4] Seto E, Yoshida M. Erasers of histone acetylation: the histone deacetylase enzymes [J]. Cold Spring Harb Perspect Biol. 2014;6(4):a018713–a.
[5] Grozinger CM, Hassig CA, Schreiber SL 1999. Three proteins define a class of human histone deacetylases related to yeast Hda1p [J]. Proc Natl Acad Sci 96: 4868–4873.
[6] Kao HY, Downes M, Ordentlich P, Evans RM 2000. Isolation of a novel histone deacetylase reveals that class I and class II deacetylases promote SMRT-mediated repression [J]. Genes Dev 14: 55–66.
[7] Kao HY, Lee CH, Komarov A, Han CC, Evans RM 2002. Isolation and characterization of mammalian HDAC10, a novel histone deacetylase [J]. J Biol Chem 277: 187–193.
[8] Frye RA 1999. Characterization of five human cDNAs with homology to the yeast SIR2 gene: Sir2-like proteins (sirtuins) metabolize NAD and may have protein ADP-ribosyltransferase activity [J]. Biochem Biophys Res Commun 260: 273–279.
[9] Du J, Zhou Y, et al. 2011. Sirt5 is a NAD-dependent protein lysine demalonylase and desuccinylase [J]. Science 334: 806–809.
[10] Gao L, Cueto MA, Asselbergs F, Atadja P 2002. Cloning and functional characterization of HDAC11, a novel member of the human histone deacetylase family [J]. J Biol Chem 277: 25748–25755.
[11] Seto E, Yoshida M. Erasers of histone acetylation: the histone deacetylase enzymes [J]. Cold Spring Harb Perspect Biol. 2014 Apr 1;6(4):a018713.
[12] Hu E, Chen Z, et al. 2000. Cloning and characterization of a novel human class I histone deacetylase that functions as a transcription repressor [J]. J Biol Chem 275: 15254–15264.
[13] Kailin Yan, Qiang Cao, et al.Histone Deacetylase 9 Deficiency Protects against Effector T Cell-mediated Systemic Autoimmunity [J]. Immunology, Volume 286, Issue 33, 19 August 2011, Pages 28833-28843.
[14] Li Y, Peng L, Seto E. Histone Deacetylase 10 Regulates the Cell Cycle G2/M Phase Transition via a Novel Let-7-HMGA2-Cyclin A2 Pathway [J].






