蛋白质翻译后修饰(PTM)
日期:2024-01-31 09:02:59
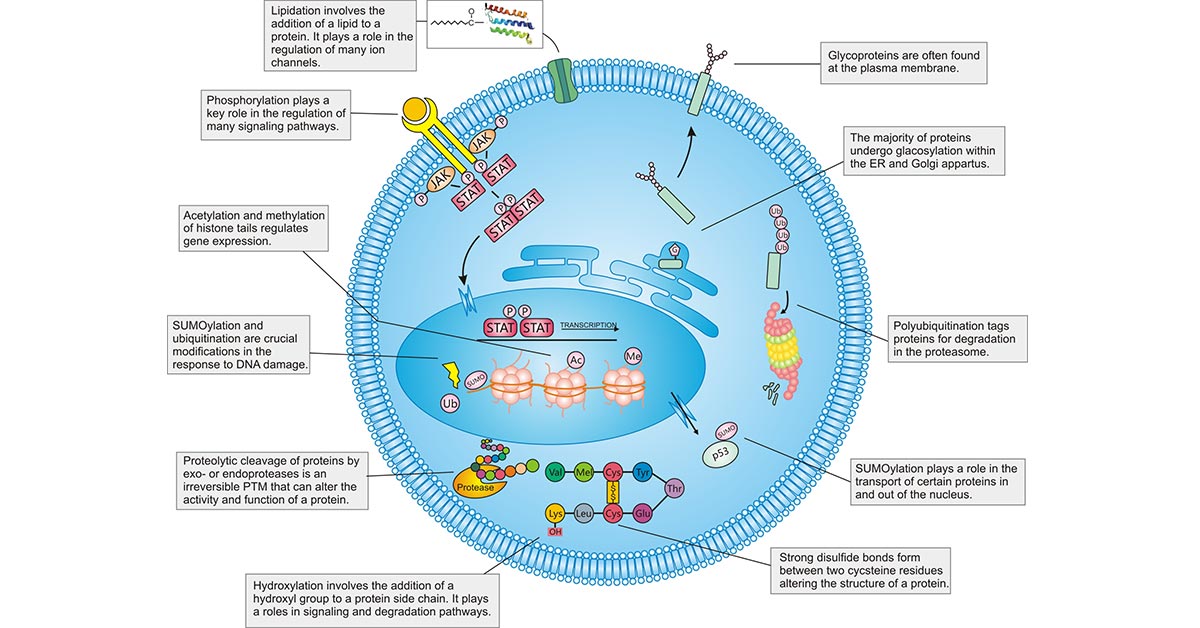
人类基因组由 20,000 到 25,000 个编码蛋白质的基因组成 [1],而人类蛋白质组估计多达 1,000,000个蛋白质 [2]。为什么人类蛋白质组的规模远远大于人类基因集?除了 mRNA 的替代剪接是蛋白质多样性的来源之外,蛋白质的翻译后修饰(PTM)也进一步影响并增加了蛋白质的变异性和复杂性。
根据生物学的 "中心法则",DNA首先转录为RNA,然后翻译成蛋白质。然而,大多数蛋白质在生物合成后还需要经过一些额外的步骤,这些步骤是细胞、组织和生物体实现其生物学功能和多样性所必需的。蛋白质翻译后的这些酶修饰称为PTM。
1. 什么是翻译后修饰?
在生物合成之后,蛋白质会通过各种方式进一步调整,以完善其结构、指定其空间方向或调节其活性。翻译后修饰(PTMs)是指通过蛋白水解裂解以及在单个或多个氨基酸上添加功能基团(如乙酰基、磷酸基和糖基)来改变蛋白质特性的这些动态的、紧密协调的加工事件 [3]。PTMs 经常出现在分泌蛋白、膜蛋白、组蛋白和其他具有重要结构/功能的蛋白质中。它们出现在细胞核、细胞质、内质网和高尔基体等细胞器中 [4]。PTMs 要么是共价修饰等可逆反应,要么是蛋白水解修饰等不可逆反应。
据估计,执行各种PTM的酶占整个蛋白质组蛋白质的5%。这些酶包括激酶、磷酸酶、转移酶和连接酶,它们可在氨基酸侧链上添加或去除功能基团、蛋白质、脂质或糖,还包括蛋白酶,它们可从氨基酸侧链上去除特定序列或调节亚基。
2. 翻译后修饰的分类
目前已发现400多种不同形式的PTM。它们可根据被修饰氨基酸侧链的类型、修饰酶的类别和可逆性程度进行分类。研究最多的 PTM 包括磷酸化、乙酰化、泛素化、甲基化、糖基化、SUMO化、脂化(棕榈酰化、肉豆蔻酰化和前酰化)和硫化。
表1:不同 PTM 的机制和功能
| PTMs的类型 | 机制 | 靶向位置 | 功能 | |||
|---|---|---|---|---|---|---|
| 磷酸化 | 可逆 | 激酶将磷酸基团从三磷酸腺苷转移到受体残基上 | 主要发生在目标蛋白质的 Ser、Thr、Tyr 和 His 残基上,也可见于 Pro、Arg、Asp 和 Cys 残基上。 | 参与复制、转录、环境应激反应、细胞运动、细胞代谢、细胞凋亡和免疫反应等关键细胞过程。 | ||
| 乙酰化 | Nα- 乙酰化 | 不可逆 | 乙酰转移酶使用乙酰 CoA 作为辅助因子,将乙酰基(COCH3)添加到赖氨酸侧链的ε-氨基上。 | 报告较多的是赖氨酸残基,但也发生在赖氨酸、Ala、Arg、Asp、Cys、Gly、Glu、Met、Pro、Ser、Thr 和 Val 残基上。 | 在染色质稳定性、蛋白质-蛋白质相互作用、细胞周期控制、细胞代谢、核运输和肌动蛋白成核等生物过程中发挥重要作用 [5] [6]。 | |
| Nε-乙酰化 | 可逆 | |||||
| O-乙酰化 | 可逆 | |||||
| 泛素化 | 可逆 | 活性泛素蛋白的 C 端与蛋白赖氨酸残基的 Nε 之间发生共价键合。 | 出现在所有 20 个氨基酸上,但更多出现在赖氨酸上。 | 在所有组织中主要通过泛素-蛋白酶体途径降解细胞内蛋白质。 | ||
| 甲基化 | 可逆 | 在目标蛋白质的赖氨酸或精氨酸残基上添加甲基。 | 发生在目标蛋白质的 Lys、Arg、Ala、Asn、Asp、Cys、Gly、Glu、Gln、His、Leu、Met、Phe 和 Pro 残基上。 | 最常见于组蛋白修饰。与各种生物过程的微调有关,从转录调控到通过异染色质组装的表观遗传沉默。 | ||
| 糖基化 | N-糖基化 | 可逆 | N 连接的聚糖与天冬酰胺或精氨酸侧链的氮相连。 | 较常出现在蛋白质和脂蛋白中的 Ser、Thr、Asn 和 Trp 残基上,但也会出现在 Ala、Arg、Asp、Ile、Lys、Val、Glu、Pro、Tyr、Cys 和 Gly 残基上。 | 参与细胞粘附、细胞-细胞和细胞-基质相互作用、分子贩运、受体激活、蛋白质溶解度效应、蛋白质折叠和信号转导、蛋白质降解以及蛋白质胞内贩运和分泌 [7-9]。 | |
| O-糖基化 | O 键聚糖连接到丝氨酸、苏氨酸、酪氨酸、羟基赖氨酸或羟基脯氨酸侧链的羟基氧上,或连接到神经酰胺等脂质上的氧原子上。 | |||||
| C-糖基化 | 色氨酸侧链的碳上添加了糖。 | |||||
| 磷酸-糖基化 | 磷酸聚糖通过磷酸丝氨酸的磷酸盐连接。 | |||||
| 糖化 | 引入 GPI 锚点,通过糖链将蛋白质与脂质连接起来。 | |||||
| SUMO酰化 | 可逆 | SUMO(SUMO1、SUMO2和SUMO3)通过三种酶,即SAE1(激活)、UBC9(共轭)和连接酶PIAS1,以共价连接的方式与底物蛋白质中的赖氨酸残基相连。 | 赖氨酸残基 | 是维持细胞中基因组完整性、转录调控、基因表达和信号转导的必要条件 [10]。调控 DNA 损伤修复、免疫反应、致癌、细胞周期进展和细胞凋亡。 | ||
| 脂化 | 棕榈酰化 | 可逆 | 通过硫酯连接将棕榈酸酯与半胱氨酸进行共价加成。 | 发生在 Cys、Gly、Ser、Thr 和 Lys 残基上。 | 在蛋白质功能调控、蛋白质-蛋白质相互作用、膜-蛋白质关联、神经元发育、信号转导、细胞凋亡和有丝分裂中发挥关键作用 [11]。 | |
| 肉豆蔻酰化 | 不可逆 | 肉豆蔻酸通过共价键与 N 端甘氨酸残基相连。 | 在甘氨酸上出现的频率较高,而在赖氨酸上出现的频率较低。 | 调节蛋白质结构的成熟、信号传递、细胞外通讯、新陈代谢和酶的催化活性。 | ||
| 异戊烯酰化 | 法呢酰化 | 不可逆 | 法呢酰焦磷酸加到半胱氨酸残基上。 | 出现在半胱氨酸和底物蛋白质的羧基末端附近。 | 促进蛋白质与蛋白质之间的相互作用、内吞调节、细胞生长、分化、增殖和蛋白质贩运。 | |
| 麦角酰化 | 不可逆 | 在半胱氨酸残基上添加焦磷酸香叶酯。 | ||||
| 硫化 | 不可逆 | TPST1 或 TPST2 可将活化的硫酸盐从 3-磷酸腺苷-5-磷酸硫酸盐转移到多肽酸性基团内的酪氨酸残基上 | 发生在目标蛋白质的 Tyr、Cys 和 Ser 上。 | 在蛋白质与蛋白质之间的相互作用、白细胞在内皮细胞上的滚动、视觉功能和病毒进入细胞等方面起着至关重要的作用。 | ||
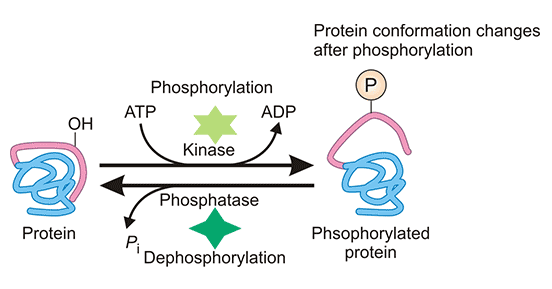
图2. 蛋白质的磷酸化
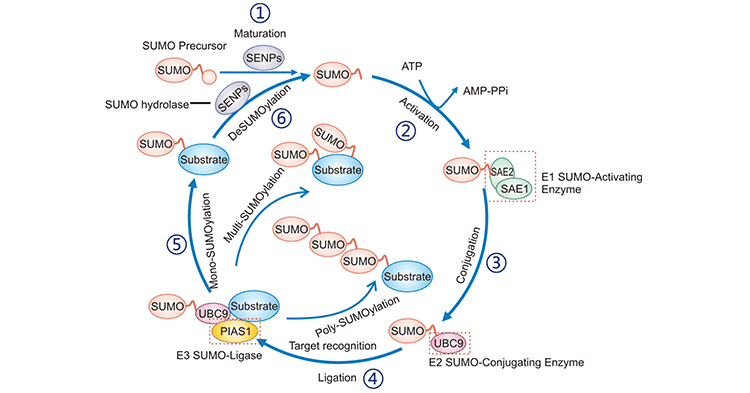
图3. SUMO化催化循环
图片来源:https://www.caister.com/cimb/v/v40/189.pdf
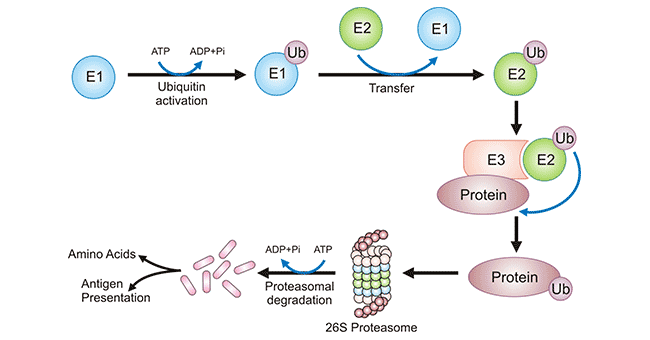
图4. 蛋白质的泛素化过程及其降解
图片来源:https://www.frontiersin.org/articles/10.3389/fmicb.2018.02738/full
3. 翻译后修饰的生物学意义
蛋白质的翻译后修饰(PTMs)在生物体内发挥着重要作用,它使蛋白质结构更加复杂,蛋白质功能更加完善,调控更加精细,蛋白质的作用更加特异。PTM通过拓宽修饰氨基酸残基的结构和理化性质范围,扩大了蛋白质结构的可变性和多样性,并引入了新的功能 [12]。由于不同的PTM可导致蛋白质理化性质的不同改变 [13],因此不同的修饰可赋予同一蛋白质不同的功能。一个蛋白质可以有不同种类和数量的PTM。
PTM 对蛋白质功能的影响是多方面的。它们可能会影响酶的功能和组装 [14]、蛋白质的活性、蛋白质的寿命、蛋白质与蛋白质之间的相互作用 [15]、细胞-细胞/基质之间的相互作用、分子迁移、受体活化、蛋白质的可溶性 [16-18]、蛋白质的折叠 [19]、蛋白质的位置、蛋白质的稳定性和蛋白质的亲电性等蛋白质的行为和特性。因此,这些修饰在多种生物过程中发挥作用,包括信号转导 [20] [21]、基因表达调控、DNA修复、细胞存活和细胞周期控制。
4. 翻译后修饰的检测
检测翻译后修饰,了解它们如何发挥作用、影响蛋白质组和控制基因组,将极大地推动我们对遗传学和表观遗传学的理解。PTM 鉴定对于阐明复杂细胞过程和疾病的机理非常重要。检测 PTM 的实验方法有很多,包括 Western 印迹(WB)、反相蛋白质阵列(RPA)、基于免疫沉淀(IP)的方法、质谱(MS)、体外检测和免疫荧光。这些方法在易用性、复杂性、成本和结果交付方面存在很大差异。可以将两种或多种方法结合起来,对感兴趣的蛋白质的 PTM 进行鉴定、验证和机理表征。
表2: 不同PTM检测方法之间的差异
| 方法 | 原理 | 优势 | 劣势 |
|---|---|---|---|
| 免疫印记(Western blotting,WB) | 在丙烯酰胺凝胶中根据分子量从复杂的细胞裂解液中分离出不同的蛋白质,然后将分离后的蛋白质转移到膜上,用针对所选 PTM 的一抗和二抗进行印迹。 | 可研究 PTM 的内源性改变。无需特定工具。 | 需要针对目标蛋白质的特异性抗体。没有位点特异性。由于 PTM 修饰可能会阻断目标蛋白质的抗体结合位点,因此可能出现假阴性。 |
| 反相蛋白质阵列(RPA) | 使用一种将细胞裂解液固定在小点上的平台,允许特异性识别相关 PTM 的抗体对预选蛋白质的 PTM 进行定量。 | 这是一种基于抗体的灵敏蛋白质组学方法,可同时对多个样本集的多种蛋白质和翻译后修饰进行定量。相对较高的重现性、灵敏度和稳健性。 | 需要事先筛选抗体并验证其特异性。收集样本和打印粘性样本的能力是 RPA 技术的一个局限。 |
| 基于免疫沉淀 (IP) 的方法 | 固定在固体支持基质(如琼脂糖树脂)上的抗体与相关蛋白质结合,而复合裂解物中的其他蛋白质则被洗去。使用洗脱缓冲液洗脱捕获的蛋白质,并将其浓缩分离。分离出的蛋白质可进一步进行 WB 或 MS 分析。 | 适用于富集感兴趣的目标蛋白质上特定的、低丰度的 PTM。经过优化的高质量 IP 试剂可提高结果的准确性。 | 进一步分析需要其他方法,如 WB 或 MS。 |
| 质谱分析法(MS) | 用特定的蛋白酶(通常是胰蛋白酶)消化感兴趣的蛋白质裂解物,富集特定的 PTM,然后用 LC-MS/MS 进行分析。收集数据后,利用计算算法识别肽和蛋白质及其相对定量。用于检测 PTMs 底物和绘制 PTMs 位点图。 | 可检测大量修饰蛋白质,灵敏度和特异性高。 | 耗时且具有高度的 PTM 特异性。需要质谱仪和熟悉操作仪器的相关专家。质谱仪的局限性和偏差。 |
| 免疫荧光(IF) | 可用于研究组织或细胞中 PTM 特征的整体和空间变化。可能是一种有用的生物读数。 | 可用于检查蛋白质在整个细胞中的定位,并确定蛋白质在不同细胞条件下的特定形式的定位。 | 不能用于鉴定特定目标的 PTM。 |
5. 翻译后修饰对疾病的影响
翻译后修饰几乎影响正常细胞生物学的所有方面,并对各种分子功能进行微调。PTMs 的失调可导致细胞生长、代谢、分化和凋亡等重要生物过程的功能障碍,从而导致疾病的发生和发展。因此,识别和了解 PTMs 对细胞生物学研究、疾病治疗和预防至关重要。
磷酸化途径的紊乱与癌症、心脏病、阿尔茨海默病和帕金森病等疾病有关 [22]。SUMOylation 会影响癌细胞信号传导和基因网络,从而调节炎症、免疫和 DNA 损伤,在致癌、增殖、转移和凋亡之间建立联系。乙酰化赖氨酸失调可导致癌症、衰老、免疫问题、神经系统疾病和心血管疾病等严重疾病 [5] [23] [24]。泛素途径的功能障碍可导致多种疾病,如不同的癌症、代谢综合征、炎症性疾病、2 型糖尿病和神经退行性疾病 [25] [26]。甲基化缺陷可导致多种疾病,如癌症、智力迟钝(安吉尔曼综合征)、糖尿病、脂质沉着病和闭塞性疾病 [27] [28]。糖基化缺陷在癌症、肝硬化、糖尿病、艾滋病病毒感染、老年痴呆症和动脉粥样硬化等多种疾病的发病过程中起着重要作用 [9] [29]。棕榈酰化功能障碍与许多疾病相关,包括亨廷顿氏病、精神分裂症和阿尔茨海默病等神经系统疾病以及不同的癌症 [30-32]。前酰化紊乱是癌症、心脑血管疾病、骨病、早衰症、代谢性疾病和神经退行性疾病的发病机制之一。
参考文献:
[1] Human Genome Sequencing Consortium (2004) Finishing the euchromatic sequence of the human genome. Nature 431: 931–945.
[2] Jensen ON. Modification-specific proteomics: characterization of post-translational modifications by mass spectrometry. Curr Opin Chem Biol 2004, 8: 33–41.
[3] Ramazi, S., Allahverdi, A. and Zahiri, J. Evaluation of post-translational modifications in histone proteins: a review on histone modification defects in developmental and neurological disorders [J]. J. Biosci. 2020, 45, 135.
[4] Blom, N., Sicheritz-Pontén, T., Gupta, R. et al. Prediction of post-translational glycosylation and phosphorylation of proteins from the amino acid sequence [J]. Proteomics, 2004, 4, 1633–1649.
[5] Choudhary, C., Kumar, C., Gnad, F. et al. (2009) Lysine acetylation targets protein complexes and co-regulates major cellular functions. Science, 325, 834–840.
[6] Wellen, KE., Hatzivassiliou, G., et al. ATP-citrate lyase links cellular metabolism to histone acetylation. Science., 2009, 324, 1076–80.
[7] Haltiwanger, R.S. and Lowe, J.B. Role of glycosylation in development. Annu. Rev. Biochem., 2004, 73, 491–537.
[8] Ohtsubo, K. and Marth, J.D. Glycosylation in cellular mechanisms of health and disease. Cell, 2006, 126, 855–867.
[9] Goulabchand, R., Vincent, T., Batteux, F. et al. Impact of autoantibody glycosylation in autoimmune diseases. Autoimmun. Rev., 2014, 13, 742–750.
[10] Han, Z. J., Feng, Y. H., et al. The post-translational modification, SUMOylation, and cancer (Review). Int. J. Oncol.52, 1081–1094 (2018).
[11] Aicart-Ramos, C., Valero, R.A. and Rodriguez-Crespo, I. Protein palmitoylation and subcellular trafficking. Biochim. Biophys. Acta (BBA) Biomembr., 2011, 1808, 2981–2994.
[12] Walsh, C. T., Garneau-Tsodikova, S., and Gatto, G. J. Jr. Protein posttranslational modifications: the chemistry of proteome diversifications. Angew. Chem. Int. Ed. Engl. 2005, 44, 7342–7372.
[13] Mann, M., and Jensen, O. N. Proteomic analysis of post-translational modifications. Nat. Biotechnol. 2003, 21, 255–261.
[14] Ryšlavá, H., Doubnerová, V., Kavan, D. et al. Effect of posttranslational modifications on enzyme function and assembly. J. Proteomics, 2013, 92, 80–109.
[15] Marshall, C. Protein prenylation: a mediator of protein-protein interactions. Science, 1993, 259, 1865–1867.
[16] Haltiwanger, R.S. and Lowe, J.B. Role of glycosylation in development. Annu. Rev. Biochem., 2004, 73, 491–537.
[17] Karve, T.M. and Cheema, A.K. Small changes huge impact: the role of protein posttranslational modifications in cellular homeostasis and disease. J. Amino Acids, 2011, 1–13.
[18] Ohtsubo, K. and Marth, J.D. Glycosylation in cellular mechanisms of health and disease. Cell, 2006, 126, 855–867.
[19] Del Monte, F. and Agnetti, G. Protein post-translational modifications and misfolding: new concepts in heart failure. Proteomics Clin. Appl., 2014, 8, 534–542.
[20] Deribe YL, Pawson T, Dikic I. Post-translational modifications in signal integration. Nat Struct Mol Biol 2010, 17: 666–672.
[21] Zhao S, Xu W, et al. Regulation of cellular metabolism by protein lysine acetylation. Science 2010, 327: 1000–1004.
[22] Nsiah-Sefaa, A. and McKenzie, M. Combined defects in oxidative phosphorylation and fatty acid β-oxidation in mitochondrial disease. Biosci. Rep., 2016, 36, e00313.
[23] Falkenberg, K.J. and Johnstone, R.W. Histone deacetylases and their inhibitors in cancer, neurological diseases and immune disorders. Nat. Rev. Drug Discov., 2014, 13, 673.
[24] Park, G., Tan, J., Garcia, G. et al. Regulation of histone acetylation by autophagy in Parkinson disease. J. Biol. Chem., 2016, 291, 3531–3540.
[25] Micel, L.N., Tentler, J.J., Smith, P.G. et al. Role of ubiquitin ligases and the proteasome in oncogenesis: novel targets for anticancer therapies. J. Clin. Oncol., 2013, 31, 1231.
[26] Popovic, D., Vucic, D. and Dikic, I. Ubiquitination in disease pathogenesis and treatment. Nat. Med., 2014, 20, 1242–1253.
[27] Robertson, K.D. DNA methylation and human disease. Nat. Rev. Genet., 2005, 6, 597.
[28] Sun, G.-D., Cui, W.-P., Guo, Q.-Y. et al. Histone lysine methylation in diabetic nephropathy. J. Diabetes Res., 2014, 1–9.
[29] Lauc, G., Huffman, J.E., Pučić, M. et al. Loci associated with N-glycosylation of human immunoglobulin G show pleiotropy with autoimmune diseases and haematological cancers. PLoS Genet., 2013, 9, e1003225.
[30] Li, S., Li, J., Ning, L. et al. In silico identification of protein S-palmitoylation sites and their involvement in human inherited disease. J. Chem. Inf. Model,2015, 55, 2015–2025.
[31] Meckler, X., Roseman, J., Das, P. et al. Reduced Alzheimer's disease β-amyloid deposition in transgenic mice expressing S-palmitoylation-deficient APH1aL and nicastrin. J. Neurosci., 2010, 30, 16160–16169.
[32] Resh, M.D. Palmitoylation of proteins in cancer. Biochem. Soc. Trans., 2017, 45, 409–416.
下一篇: 蛋白质磷酸化