组蛋白ADP-核糖基化
直到20世纪90年代末和21世纪初,研究人员才开始发现组蛋白ADP-核糖基化作为一种特定修饰。研究表明,PARP酶可以ADP-核糖基化组蛋白蛋白,特别是组蛋白H1和组蛋白H2B。随后的研究表明,组蛋白ADP-核糖基化具有广泛的作用。
1. 什么是组蛋白ADP-核糖基化?
ADP-核糖基化是指将辅因子烟碱酰胺腺嘌呤二核苷酸(NAD+)中的ADP-核糖(ADPr)基团转移到组蛋白蛋白上的特定氨基酸(Lys、Arg、Glu、Asp和Ser)的过程。
ADP-核糖基化是在DNA损伤时产生的最早的损伤信号之一。组蛋白作为ADP-核糖基化的主要底物,因为它们靠近DNA [1,2]。
所有五种组蛋白蛋白都被广泛地ADP-核糖基化,尽管只占很小的百分比。组蛋白ADP-核糖基化被提出是一种共价修饰 [8],主要涉及ADP-核糖单体或链或分支聚合物 [9]。组蛋白蛋白的ADP-核糖基化模式因染色质组成而异。在天然染色质中,组蛋白H1是主要的ADP-核糖受体,比如H1E2和H1E15,而在H1耗竭的染色质中,组蛋白H2B成为ADP-核糖基化最严重的组蛋白 [10]。
2. 参与组蛋白ADP-核糖基化的组分
组蛋白ADP-核糖基化是一个由三类关键蛋白调控的动态和可逆酶法过程:写入者、擦除者和读取者。
2.1 写入者
催化组蛋白ADP-核糖基化修饰的酶是ADP-核糖基转移酶(ARTs)或多聚(ADP-核糖)聚合酶(PARPs),也被称为ADP-核糖“写入者”,它们分别具有单核苷酸或多聚ADP-核糖转移酶活性。
有三个家族的ADP-核糖基转移酶,包括具有单和多ADP-核糖转移酶活性的白喉毒素样ADP-核糖基转移酶(ARTDs,以前被称为PARPs),肠毒素样ADP-核糖基转移酶(ARTCs)和Sir2家族的NAD+依赖性蛋白去乙酰化酶(去乙酰化酶)。后两者仅作为单ADP-核糖基转移酶发挥作用。
| ADP-核糖转移酶 | 亚型 | 细胞亚结构定位 | 酶活性 |
|---|---|---|---|
| ARTDs | ARTD1 | Nucleus | Poly-ADP-核糖转移酶 |
| ARTD2 | Nucleus | Poly-ADP-核糖转移酶 | |
| ARTD3 | Nucleus | Mono/Poly-ADP-核糖转移酶 | |
| ARTD4 | Nucleus/cytoplasm | Poly-ADP-核糖转移酶 | |
| ARTD5 | Nucleus/cytoplasm | Poly-ADP-核糖转移酶 | |
| ARTD6 | Nucleus/cytoplasm | Poly-ADP-核糖转移酶 | |
| ARTD7 | Nucleus | Mono/Poly-ADP-核糖转移酶 | |
| ARTD8 | Nucleus/cytoplasm | Mono/Poly-ADP-核糖转移酶 | |
| ARTD9 | Nucleus/cytoplasm | Mono/Poly-ADP-核糖转移酶 | |
| ARTD10 | Nucleus/cytoplasm | Mono/Poly-ADP-核糖转移酶 | |
| ARTD11 | - | Mono/Poly-ADP-核糖转移酶 | |
| ARTD12 | Nucleus | Mono/Poly-ADP-核糖转移酶 | |
| ARTD13 | Nucleus | Catalytically inactive | |
| ARTD14 | Nucleus | 核糖转移酶 | |
| ARTD15 | Nucleus/cytoplasm | 核糖转移酶 | |
| ARTD16 | - | 核糖转移酶 | |
| ARTD17 | - | 核糖转移酶 | |
| ARTCs | ARTC1 | Ecto-cellular | - |
| ARTC2 | Ecto-cellular | - | |
| ARTC3 | Ecto-cellular | - | |
| ARTC4 | Ecto-cellular | - | |
| ARTC5 | Ecto-cellular | - | |
| Special ARTs | ADPRT1a | - | 核糖转移酶 |
| ADPRT2 | - | 核糖转移酶 | |
| Sirtuins | SIRT1 | Nucleus | - |
| SIRT2 | Nucleus/Cytoplasm | - | |
| SIRT3 | Mitochondria | - | |
| SIRT4 | Mitochondria | 核糖转移酶 | |
| SIRT5 | Mitochondria | - | |
| SIRT6 | Nucleus | 核糖转移酶 | |
| SIRT7 | Nucleus (nucleoli) | 核糖转移酶 |
表格信息来源: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8027728/
| 底物 | 修饰的氨基酸 | 酶 | 反应效果 |
|---|---|---|---|
| H1 | E2, E14, E116 and K213 | - | H1-H1相互作用的调控 |
| R34 | - | 阻止cAMP依赖的组蛋白H1的磷酸化 | |
| Q/N | ARTD3 | DNA修复 | |
| H2A | 未知 | Sir2 | 对氧化应激/DNA损伤的反应 |
| 未知 | Sir2 | 抑制组蛋白乙酰化/沉默染色质区域 | |
| R/E | - | 未知 | |
| K13 | ARTD1 | 未知 | |
| H2B | E2 | ARTD3/ARTD10 | 未知 |
| E18/E19 | ARTs, Adprt1a/Adprt2 | 对氧化应激/DNA损伤的反应 | |
| 未知 | Sir2 | 抑制组蛋白乙酰化/沉默染色质区域 | |
| K30 | ARTD1 | 未知 | |
| H3 | 未知 | SIRT6, Sir2 | 抑制组蛋白乙酰化/沉默染色质区域 |
| R | - | 细胞增殖 | |
| K27 | ARTD1 | 未知 | |
| K37 | ARTD1 | 未知 | |
| H4 | R | Sir2 | 合成后修饰,包括核心组蛋白的乙酰化 |
| 未知 | Sir2 | 对氧化应激/DNA损伤的反应 | |
| 未知 | ARTD10 | 未知 | |
| 未知 | Sir2-relatedprotein | 抑制组蛋白乙酰化 | |
| K16 | ARTD1 | 未知 |
表格信息来源: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8027728/
(Note: E, glutamate; K, lysine; R, arginine; Q, glutamine; N, asparagine)
2.2 擦除者
末端ADP-核糖基水解酶(TARG)和多聚ADP-核糖葡萄糖水解酶(PARG),也被称为“擦除者”,分别负责从ADP-核糖化的组蛋白上移除ADP-核糖单体或聚合物。
| 擦除酶 | 蛋白底物 |
|---|---|
| PARG | PAR-protein |
| TARG1 | PAR-Glu, MAR-Asp/Glu |
| MacroD1 | MAR-Asp/Glu |
| MacroD2 | MAR-Asp/Glu |
| ARH1 | MAR-Arg |
| ARH3 | PAR-protein MAR-Ser |
| NUDT9 | PAR-protein |
| NUDT16 | PAR-protein, MAR-protein |
| ENPP1 | PAR-protein, MAR-protein |
2.3 读取者
ADP-核糖修饰被含有聚ADP-核糖结合锌指(PBZ)结构域或宏域的蛋白识别,随后调节染色质结构和转录。
| 读取模块 | 组蛋白 | 功能 |
|---|---|---|
| PBM | Histone H2A, Histone H2B, Histone H3, Histone H4 | DNA修复、染色质重排、转录、基因表达 |
| Macrodomain | macroH2A1.1 | 染色质重塑、DNA修复 |
3. Histone ADP-核糖化的机制
Histone ADP-核糖化涉及一系列复杂的分子事件。以下是该过程的简化概述:
3.1 识别DNA损伤
Histone ADP-核糖化在DNA修复中的作用是最为研究的。当细胞检测到DNA损伤,比如断裂或损伤,PARP酶会被激活,然后结合到受损DNA上,并开始在附近的组蛋白上合成ADP-核糖链。
3.2 染色质松弛
在组蛋白上添加ADP-核糖会导致染色质松弛,使受损DNA更容易被修复机制接触到。这一步骤对于高效的DNA修复至关重要。
3.3 招募修复蛋白
ADP-核糖链充当DNA修复蛋白在损伤位点组装的信号。这些蛋白随后进行必要的修复,确保基因组的完整性。
4. Histone ADP-核糖化的功能
许多研究已经证明了组蛋白多ADP-核糖化在DNA修复和DNA复制中的重要贡献 [2, 3],以及在细胞和肿瘤的增殖中的作用 [4]。组蛋白的单ADP-核糖化也有助于转录和染色质重塑 [7]。
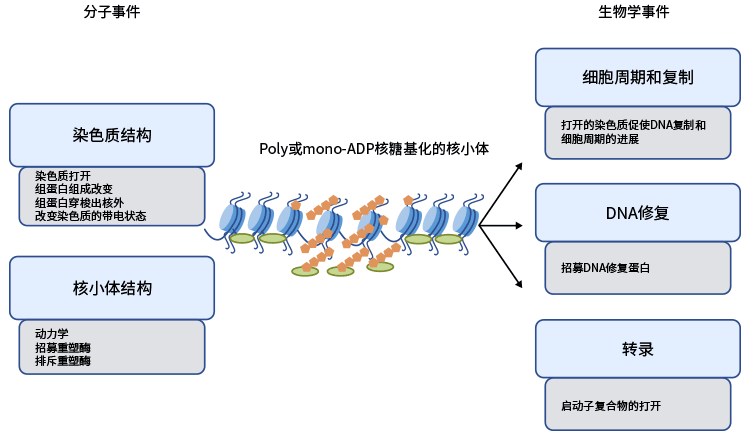
图 1. 组蛋白ADP核糖基化功能
该图片引自:https://www.sciencedirect.com/science/article/abs/pii/S0962892411001061
5. 组蛋白ADP核糖基化与其他组蛋白修饰之间的相互作用
各种PTM之间的相互作用可以通过直接竞争受体位点或间接改变修饰酶的染色质可及性而发生。特定赖氨酸残基可作为ADP-核糖的受体的发现是值得注意的,因为这些氨基酸残基也是乙酰化和甲基化的潜在靶点[5]。因此,ADP-核糖化、乙酰化、甲基化和磷酸化之间的受体位点的竞争很可能导致相互作用[3]。这一观点得到了支持,研究发现H4 K16ac在体外抑制了ADP-核糖基化[6],暗示着在体内存在类似的相互作用。
参考文献:
[1] Ogata N, Ueda K, and Hayaishi O (1980) ADP-ribosylation of histone H2B-identification of glutamic acid residue 2 as the modification site [J]. J. Biol. Chem 255, 7610–7615.
[2] Messner S, Hottiger MO (2011) Histone ADP-ribosylation in DNA repair, replication and transcription [J]. Trends Cell Biol 21: 534–542.
[3] Hottiger MO. ADP-ribosylation of histones by ARTD1: an additional module of the histone code [J]? FEBS Lett. 2011; 585:1595–99.
[4] Boulikas T, Bastin B, Boulikas P, Dupuis G. Increase in histone poly (ADP-ribosylation) in mitogen-activated lymphoid cells [J]. Exp Cell Res. 1990; 187:77–84.
[5] Kouzarides T. Chromatin modifications and their function. Cell. 2007;128:693–705.
[6] Messner S, Altmeyer M, et al. PARP1 ADP-ribosylates lysine residues of the core histone tails. Nucleic Acids Res. 2010;38:6350–6362.
[7] Boulikas T. Relation between carcinogenesis, chromatin structure and poly(ADP-ribosylation) (review) [J]. Anticancer Res. 1991;11:489–527.
[8] P. Stone, W. Lorimer, W. Kidwell. Properties of the complex between histone H1 and poly(ADP-ribose synthesised in HeLa cell nuclei [J]. Eur. J. Biochem., 81 (1977), pp. 9-18.
[9] L. Burzio, P. Riquelme, S. Koide. ADP ribosylation of rat liver nucleosomal core histones [J]. J. Biol. Chem., 254 (1979), pp. 3029-3037.
[10] A. Huletsky, G. de Murcia, et al. The effect of poly(ADP-ribosyl)ation on native and H1-depleted chromatin. A role of poly(ADP-ribosyl)ation on core nucleosome structure [J]. J. Biol. Chem., 264 (1989), pp. 8878-8886.






